Résumé
Une attention considérable a été accordée récemment pour améliorer la qualité des données dans les technologies de criblage à haut débit (HTS) largement utilisées dans la recherche sur le développement de médicaments et la toxicité chimique. Cependant, plusieurs biais spatiaux induits par l'environnement et la procédure dans les criblages expérimentaux HTS diminuent la précision de la mesure, entraînant une augmentation du nombre de faux positifs et de faux négatifs dans la sélection des « hits ». Le logiciel « HTS_corrector » a été développé pour la correction de ces biais.
L’évaluation de la performance des méthodes de correction (implémentée dans le logiciel « HTS_Corrector ») avec des données réelles affectées par un biais spatial a permis de mettre en évidence le nombre de hits.
Les méthodes pour éliminer le biais spatial et le protocole de correction de données sont efficaces pour détecter et nettoyer les données expérimentales générées par les technologies de criblage. La correction par puits semble être la méthode qui a minimisé les faux positifs et faux négatifs. Elle peut donc contribuer au processus de la recherche de nouveaux médicaments.
Introduction
Le criblage à haut débit (« high throuput screening » en anglais, ou HTS) est une méthode très utilisé dans l’industrie pharmaceutique et dans les laboratoires spécialisés, car il permet d'examiner des centaines de milliers de composés chimiques et de générer de nombreuses données expérimentales (Lachmann et al., 2016). Leurs analyses nécessitent cependant l’utilisation de méthodes statistiques et des protocoles appropriés pour détecter les candidats potentiels (appelés hits) aux médicaments (Birmingham et al., 2009 ; Malo et al., 2006). En effet, la plupart des maladies rares n’ont pas de traitement. Pour accélérer la découverte de futurs médicaments, le chercheur utilise des robots pour tester de manière précise et rapide des milliers de molécules ciblant la maladie rare qu’il étudie. Cette technique de criblage est réalisée dans des laboratoires spécialisés qu’on appelle plateforme. Ces plateformes utilisent des outils de haute technologie et de dizaine de millier de molécules référencés dans les chimiothèques. Pour effectuer un test de criblage à haut débit (HTS), les cellules vivantes, mimant la maladie à étudier, sont déposées dans les puits d’une plaque. Un robot distribue ensuite une molécule différente dans chaque puit. Ce premier test permet de repérer des molécules qui ont un intérêt thérapeutique selon des critères définis au préalable par le chercheur. Ces molécules prometteuses sont sélectionnées et passent ensuite d’autres tests pour devenir d’éventuels médicaments.
Cependant, le biais spatial au sein des plaques (biais positionnel ou erreur systématique) reste l'un des principaux obstacles des campagnes de criblage expérimentales. Ce biais peut être causée par de facteurs techniques ou environnementaux (Heyse, 2002, Makarenkov et al., 2007) et est considéré comme une évidente source de sous-estimation ou surestimation systématique de mesures de criblage spécifiques (Kevorkov et Makarenkov, 2005). Ces erreurs sont sources de production de faux positifs (composés inactifs incorrectement identifiés comme des hits) et de faux négatifs (composés actifs non détectés) dans les technologies de criblage (Birmingham et al., 2009). Typiquement, le biais spatial affecte soit les composés placés dans le même puits, la rangée ou la colonne sur toutes les plaques du test (erreur spécifique au test) ou les composés d'une rangée ou colonne spécifique d'une plaque donnée (erreur spécifique à la plaque) (Dragiev et al., 2012).
Dans le présent travail, nous nous sommes proposés dans un premier temps de résumer les divers types d’erreur et leurs méthodes de correction statistiques, pour ensuite analyser et corriger les données mises à disposition à l’aide logiciel HTS corrector.
Méthode
1. Les types d’erreurs rencontrer dans le criblage haut débit
Bien que qu’il ait eu beaucoup d’amélioration de l’automatisation du criblage à haut débit, cette méthode n’est pas à l’abri des erreurs au sein des résultats qui peuvent être surestimé ou sous-estimé et ainsi donner de faux positifs ou de faux négatifs. Les erreurs obtenues sont de deux types : les erreurs aléatoires et les erreurs systématiques.
Dans le premier cas, les erreurs aléatoires sont simplement dû au hasard et ne peuvent pas vraiment être corrigées à moins de dupliquer l’expérience pour en atténuer les effets. Ce genre de pratique n’est généralement pas faites, car trop onéreuse.
Ce qui nous intéresse plutôt ici, ce sont les erreurs systématiques que l’on définit comme étant des mesures qui sous-estiment ou surestiment l’effet d’un composant étudié, et ce, sur la même plaque ou au même endroit dans l’expérience. Des facteurs technologiques ou environnementaux peuvent être à l’origine de ces erreurs. Cela peut venir d’une erreur de la machine utilisée, une erreur de lecteur, un mauvais fonctionnement des pipettes, une différence de concentration relié à l’évaporation de l’agent utilisé, des variations dans le temps d’incubation ou de températures, ou même des irrégularités de luminosité ou de flux d’air ambiant (Caraus et al., 2015).
Il existe deux types d’erreurs systématiques qui ont des positions spécifiques sur les plaques. Le premier est le « batch effect » (que l’on pourrait traduire par « effet de traitement par lot ») qui consiste en une erreur qui apparaît dans un lot, mais pas dans un autre.
L’autre types d’erreur est le « edge effect » (appelé aussi « border effect », soit l’« effet de bordure ») qui consiste en une surestimation ou une sous-estimation des mesures situées sur les bords du plateau.
À l’origine, le biais des erreurs spécifiques des plateaux était comme suit :
![]()
Avec :
![]() comme étant la valeur biaisée dans le puit (i,j) du plateau p.
comme étant la valeur biaisée dans le puit (i,j) du plateau p.
![]() est la valeur non biaisée dans ce puit
est la valeur non biaisée dans ce puit
![]() est l’erreur affectant la ligne i du plateau p
est l’erreur affectant la ligne i du plateau p
![]() est l’erreur affectant la colonne j du plateau p
est l’erreur affectant la colonne j du plateau p
![]() est l’erreur aléatoire affectant le puit (i,j) du plateau p
est l’erreur aléatoire affectant le puit (i,j) du plateau p
![]()
Comme ces erreurs se répètent au même endroit sur les plaques, il est alors possible de les repérer et de les quantifier pour ensuite les corriger par des méthodes statistiques.
2. Les méthodes de correction statistiques
Avant d’utiliser des méthodes statistiques pour corriger les données des plateaux, il est important de les normaliser afin de pouvoir mieux les comparer. On utilise alors un Z-score dont la formule est la suivante :
![]()
Suite à ça, il est alors possible d’utiliser les méthodes de corrections que nous avons vues, qui sont la soustraction de l’arrière-plan (« background substraction ») et la correction par puit (« well correction »). D’autres méthodes existent, tel que le « median polish » et le B-score, mais nous ne développerons pas ces dernières dans le présent rapport.
2.1 La soustraction de l’arrière-plan
L’arrière-plan constitue en quelque sorte le bruit de fond qu’on peut retrouver sur les plaques de criblage.
Pour évaluer cet arrière-plan, on utilise la formule suivante :
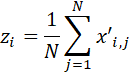
où :
![]() est la valeur normalisée dans le puit i du plateau j,
est la valeur normalisée dans le puit i du plateau j,
![]() est la valeur d’arrière-plan du puit i,
est la valeur d’arrière-plan du puit i,
![]() est le nombre total de plateaux.
est le nombre total de plateaux.
Ainsi, les étapes pour soustraire l’arrière-plan sont les suivantes :
- Normalisation des données par plateau
- Élimination des données aberrantes (il est important de le faire avant d’évaluer l’arrière-plan, car nous ne voulons pas intégrer ces données dans le calcul)
- Calcul de l’arrière-plan
- Élimination des erreurs systématiques en soustrayant l’arrière-plan des données normalisées
- Sélection des données d’intérêt (« hit selection »)
Il est possible de soustraire directement l’arrière-plan des données brut, ou bien de soustraire une approximation de celui-ci. Cette approximation est obtenue par un polynôme de quatrième degré.
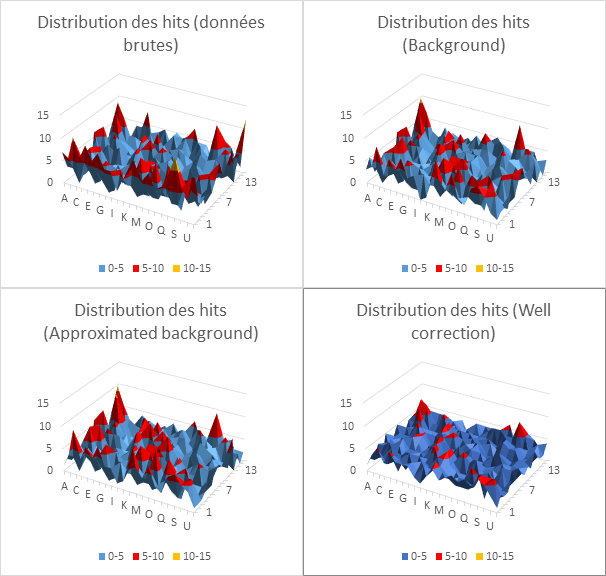
Dans l’illustration suivante créée grâce au logiciel HTS Corrector (dont nous allons parler plus loin), nous voyons bien l’effet de bordure en bleu qui sont en fait des données qui ont été sous-estimées et qui vont être corrigées lorsqu’elles seront soustraites des données brutes.
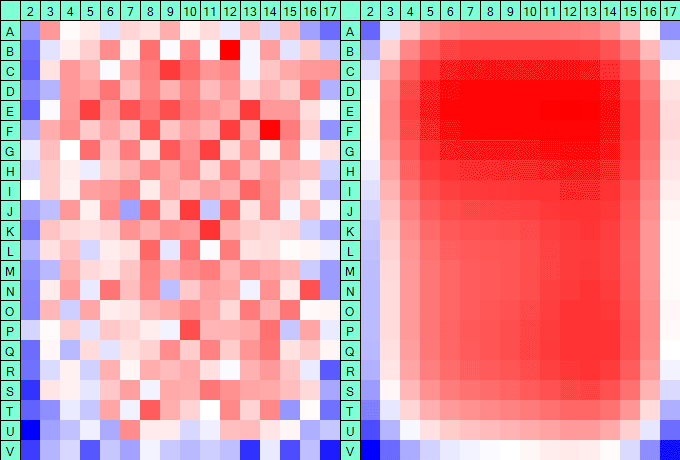
Grâce au logiciel HTS Corrector, il est possible de récupérer les données sous forme d’un tableau que l’on peut ensuite entrer dans Excel. Cela nous permet de créer des diagrammes en trois dimension qui nous donnent une meilleure idée de l’arrière-plan.
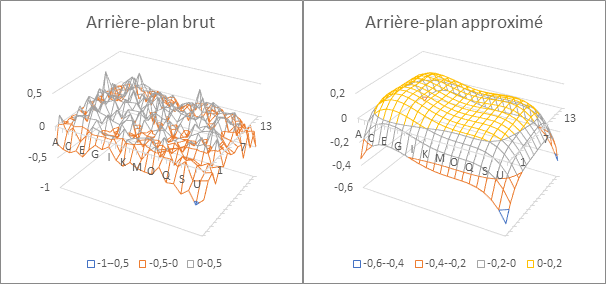
Nous verrons concrètement comment appliquer cette méthode lors de l’utilisation d’HTS Corrector.
2.2 La correction par puit
L’autre méthode de correction que nous avons vue est la méthode de correction par puit. Les données sont normalisées à l’intérieur de chaque puit pour le criblage au complet. On analyse ensuite ces données pour appliquer une correction à réaliser pour chaque puit grâce à une fonction linéaire ou polynomiale.
Les étapes de la correction par puit sont les suivantes :
- Normalisation des données pour chaque plateau
- Calcul de la tendance (ascendante ou descendante) pour chaque puit en utilisant une approximation linéaire ou polynomial
- Soustraction de la tendance obtenue à partir des valeurs de plateau normalisé
- Normalisation des données pour chaque puit
- Rééxamination de la surface de distribution des hits
Les méthodes présentées dans ce rapport peuvent être appliquées grâce au logiciel HTS Corrector présenté dans la partie suivante.Utilisation de HTS Corrector
3. Utilisation de HTS Corrector
HTS Corrector est un logiciel développé par le Dr. Vladimir Makarenkov et son équipe dans le but de corriger les erreurs systématiques dans les résultats de criblage à haut débit. Ce logiciel est disponible gratuitement à l’adresse suivante : http://www.info2.uqam.ca/~makarenkov_v/HTS/home.php .
Afin d’illustrer l’utilisation de ce logiciel, nous allons utiliser deux jeux de données tirés du site de HTS Corrector, soit Harvard_164 et McMaster_1250. La comparaison des données se fera avec une sélection des hits à μ-2σ.
Pour analyser les données, nous allons utiliser la répartition des hits par ligne et par colonne sous forme de diagramme en bâtons avec les corrections en superposition. La sélection des hits devrait être uniforme par ligne et par colonne s’il n’y avait pas de biais, nous aurons alors un meilleur support visuel pour comparer les données entre elles avec et sans correction.
Nous allons aussi créer des diagrammes en trois dimension de la distribution des hits afin de comparer les données entre elles.
Résultats
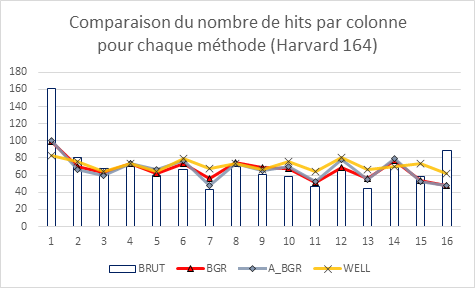
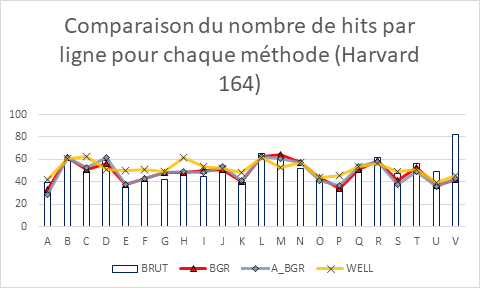
Le premier jeu de données analysées est celui de Havard 164. Comme décrit précédemment, il s’agit de lectures de 164 plateaux de 384 puits d’un essai d’inhibition de la protéine MurG.
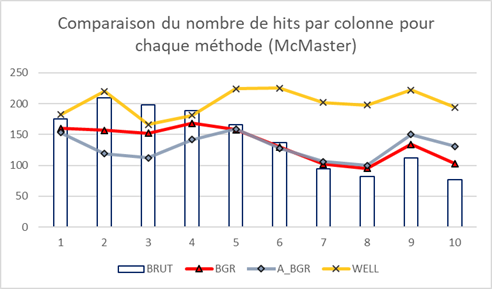
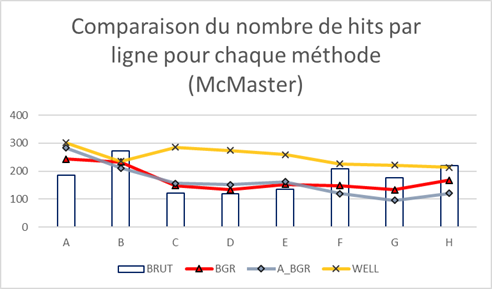
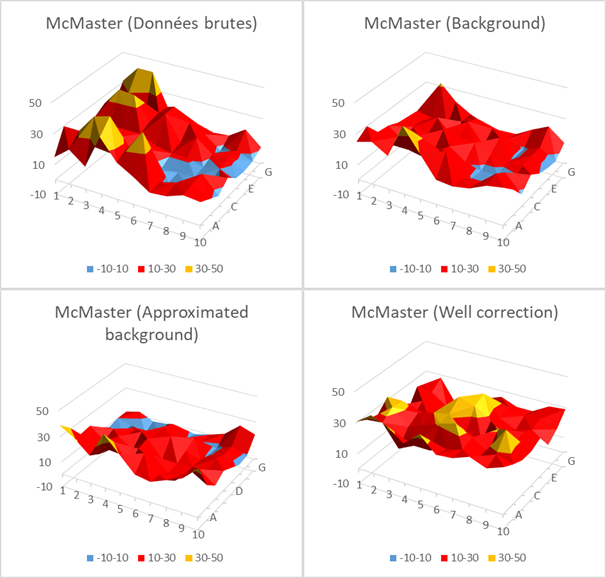
Le deuxième jeu de données analysées est celui de McMaster_1250, utilisé dans l’article de Elowe et al.
Discussion
Le but de ce rapport est de comparer les méthodes de correction des erreurs systématiques et lorsqu’on regarde les graphes des données brutes, on peut voir qu’il y a bel et bien un biais des données : dans le cas de Harvard_164, il s’agit des données sur le pourtour de la plaque, alors que pour McMaster 1250 on observe des creux et des bosses très marqués.
Les méthodes de correction par soustraction de l’arrière-plan (approximé ou non) donnent quasiment les mêmes résultats. Elles permettent de « lisser » quelque peu les données, mais restent quand même assez proches des données brutes. Cela s’observe aussi bien dans les deux jeux de données pour ce qui est des diagrammes en bâton, mais ceci est assez flagrant dans le cas des données de Harvard 164, surtout lorsqu’on observe les graphiques en trois dimensions.
Enfin, on voit vraiment une nette différence avec la méthode de correction par puit qui permet d’homogénéiser les données, et ce, dans les deux jeux de données. En effet, le biais systématique semble avoir été corrigé dans tous les types de graphe utilisés.
On peut donc dire que la méthode de correction par puit semble la plus efficace pour traiter les données de criblage à haut débit si on la compare à la soustraction de l’arrière, car elle permet une meilleure homogénéisation des données traitées.
Conclusion
Des deux méthodes vues dans ce rapport, soit la soustraction de l’arrière-plan (« background substraction ») et la correction par puit (« well correction ») servant à minimiser l'impact de l'erreur systématique dans les données HTS brutes et ainsi de rapprocher la surface de distribution d'impact d'une surface plane, il semblerait que la méthode de correction par puit soit la meilleure des deux. Elle semble donner des résultats plus précis que l'algorithme procédant par la suppression de l’arrière-plan évalué. Certes, nous avons observé cette tendance avec seulement deux jeux de données, alors il serait intéressant d’utiliser cette méthode avec d’autres données de criblage à haut débit afin d’éprouver cette méthode. De nombreuses banques existent sur internet que ce soit pour des composés chimiques, mais aussi pour de l’ADN complémentaire ou de l’ARN interférent.
Bibliographie
- Birmingham, A. et al. (2009) ‘Interference Screens’, Nature Methods, 6(8), pp. 569–575. doi: 10.1038/nmeth.1351.Statistical.
- Caraus, I. et al. (2015) ‘Detecting and overcoming systematic bias in highthroughput screening technologies: A comprehensive review of practical issues and methodological solutions’, Briefings in Bioinformatics, 16(6), pp. 974–986. doi: 10.1093/bib/bbv004.
- Dragiev, P., Nadon, R. and Makarenkov, V. (2012) ‘Two effective methods for correcting experimental high-throughput screening data’, Bioinformatics, 28(13), pp. 1775–1782. doi: 10.1093/bioinformatics/bts262.
- Elowe, N. H. et al. (2005) ‘Experimental screening of dihydrofolate reductase yields a “test set” of 50,000 small molecules for a computational data-mining and docking competition’, Journal of Biomolecular Screening, 10(7), pp. 653–657. doi: 10.1177/1087057105281173.
- Helm, J. S. et al. (2003) ‘Identification of active-site inhibitors of MurG using a generalizable, high-throughput glycosyltransferase screen’, Journal of the American Chemical Society, 125(37), pp. 11168–11169. doi: 10.1021/ja036494s.
- Kevorkov, D. and Makarenkov, V. (2005) ‘Statistical analysis of systematic errors in high-throughput screening’, Journal of Biomolecular Screening, 10(6), pp. 557–567. doi: 10.1177/1087057105276989.
- Lachmann, A. et al. (2016) ‘Detection and removal of spatial bias in multiwell assays’, Bioinformatics, 32(13), pp. 1959–1965. doi: 10.1093/bioinformatics/btw092.
- Makarenkov, V. et al. (2007) ‘An efficient method for the detection and elimination of systematic error in high-throughput screening’, Bioinformatics, 23(13), pp. 1648–1657. doi: 10.1093/bioinformatics/btm145.
- Malo, N. et al. (2006) ‘Statistical practice in high-throughput screening data analysis’, Nature Biotechnology, 24(2), pp. 167–175. doi: 10.1038/nbt1186.