UNIVERSITÉ
DU QUÉBEC À MONTRÉAL
Conservation des motifs structuraux à longue
portée dans les ARN
Présenté
par : Vladimir
Reinharz
Travail présenté à Dr.Vladimir
Makarenkov et Abdoulaye Baniré Diallo
Dans le cadre du cours :
Séminaire interdisciplinaire de bio-informatique
(BIF7002)
PAR
KHELIL
Yasmine (KHEY06589500)
MELEK Soumia (MELS25599308)
Le 15 Avril 2020
Sommaire
Introduction
Géométries des paires de bases de l'ADN et de l’ARN
Rôle de la géométrie des
ARN
Motifs structuraux d’ARN
Traitement informatique des structures
secondaires de l’ARN
L'algorithme de Nussinov (paires de bases
maximales)
Géométries
des interactions entre les nucléotides : Leontis-Westhof
annotations
Motif
structural local kink turns
Isomorphismes
Carnaval
Motif A-mineur et traduction ribosomale
Épigénétique des ARNs et les modifications
chimiques
Conclusion
Introduction
Les molécules d’acide
ribonucléique (ARN) jouent un rôle important au sein des
organismes vivants, pour la transmission de l’information
génétique et comme enzymes catalysant des réactions
biochimiques dans les cellules, l’intégration transmembranaire ou
encore l’épissage. Cette richesse fonctionnelle est due à
la structure tridimensionnelle des ARN. Comprendre leurs processus de
repliement reste donc un défi important de la biologie structurale de
nos jours.
Géométries des paires de bases de
l'ADN et de l’ARN
La propriété fondamentale des
paires de bases d’ADN « ATCG» est leur superposition (on
peut superposer le mieux ou le backbone va se placer et ça rentre exactement
à l’intérieur de la même surface).
Un de nombreux mécanismes de
régulation de l’ADN est la vérification si le double brin
est régulier ou s’il y a une paire de base qui n’est pas
fonctionnelle, et cette régularité est absolument nécessaire
et une conséquence de la géométrie des paires de bases.
D’un point de vue
géométrique les « AT » chez les ADN et les
« AU » qui est l’équivalent chez les ARNs ne
sont pas superposable, et à cause de cette propriété
automatiquement y’a plus de régularité, s’il y a une
paire de base « AU » ne peut pas être
régulière l’inverse ne peut pas remplacer AU donc il est
impossible d’avoir une longue double hélice
régulière avec les Adn, ce qui entraîne des
déformations.
Par exemple, une structure complètement
standard de l’ARN simple brin retrouve des petits morceaux de double
hélice qui commence à se retourner mais à cause du manque
de régularité ils vont se pencher un peu trop vers un
côté ou ils peuvent rester régulier. Donc il est impossible
pour un ARN d’être régulier parce que
géométriquement les paires de bases ne peuvent pas être
superposable.
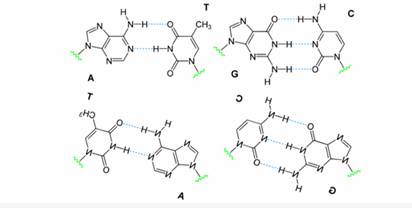

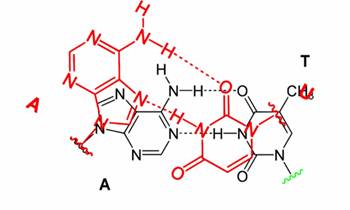
Figure 1 :
Géométries des paires de bases de l'ADN et de l’ARN
Rôle de la géométrie des
ARNs
La fonction dépend seulement de la
structure. La compréhension des liens entre la fonction et la structure
va nous permettre de :
o
Identifier les mutations
délétères
o
Concevoir de nouveaux biocapteurs (riboswitches)
et médicaments
o
Comprendre les interactions
intermoléculaires
o
Étudier l'évolution de la
vie : évolution de la complexité géométrique
Motifs structuraux d’ARN
Lors de la formation de la structure de
l’ARN, les régions appariées et non appariées
s’organisent sous forme de motifs où chacun de ces motifs est une
combinaison particulière de nucléotides appariés et non
appariés. En considérant ces motifs, l'ARN peut être alors
vu comme une molécule biologique modulaire où ses modules sont
les motifs (Leontis et al., 2006) (Hendrix et
al., 2005)
Les motifs d’ARN se distinguent en 2
classes :
●
Motifs locaux :
ce sont des motifs insérés dans les
´éléments de structure secondaire. Le terme
“local” veut dire “local à la structure secondaire”,
exemple : le Kink-turn.
●
Motifs d’interactions : ce sont des motifs de
longue-distance qui mettent en interaction deux ou plusieurs
éléments de structure secondaire distants pour induire un
repliement spatial d’une partie de la molécule (Djelloul., 2013). Ces interactions sont entre
hélices, boucles et motifs locaux, à partir de là on
distingue les réseaux d’interactions récurrent (RIN), qui
ne comprend aucune information sur les séquences, strictement sur la
topologie des interactions entre les nucléotides et leurs natures. (Reinharz et al,.
2018)

Figure 2: Motifs structuraux d’ARN.
Traitement informatique des structures
secondaires de l’ARN
Le format point-parenthèses (DBN) se
terminant par l’extension «.dbn». Il a été
popularisé par la librairie ViennaRNA spécialisée dans le
traitement des structures secondaires d’ARN et est une extension du
format FASTA, ce dernier étant utilisé pour formater les
séquences biologiques. La ligne (Figure 3) décrit les
appariements associant à chaque position des parenthèses, «
(» ou «) », indiquant que le nucléotide correspondant
à « (» est apparié au nucléotide correspondant
à «) ». Le caractère «.» indique un
nucléotide non apparié à cette position. (Jean-Pierre,2017)
GAGUUCCCGAAAGGAUGGCGGAAACGCCAGAUGCCUUGUAACCGAAAGGGGGAAUUC
((((((..((((.(((((....)))))....))))....((....))))))))
Figure 3 : Format
point-parenthèses (DBN).
L'algorithme de Nussinov (paires de bases
maximales)
Consiste à replier une séquence
d’ARN en maximisant le nombre d’appariements et qui adopte une
fonction d’énergie E qui ne prend en compte que les appariements,
pas l’empilement. Pour chaque sous-séquence allant de la position
i à j, on calcule l’énergie E (i, j) de la structure la
plus stable s (i, j) qui peut être obtenue par les 4 façons
suivantes :
2. ajouter i, non- appariés `a s (i + 1,
j).
4. réunir deux sous-structures
optimales, s (i, k) et s (k + 1, j). (Feng Lou,2012)
L’énergie E (i, j) peut donc
calculée par la récurrence suivante :
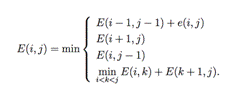
Ou e (i, j) est l’énergie de la paire de bases
i et j.
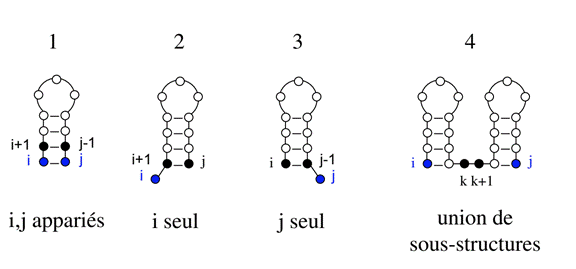
Figure 4 : Les 4 façons pour obtenir la structure S (i, j).
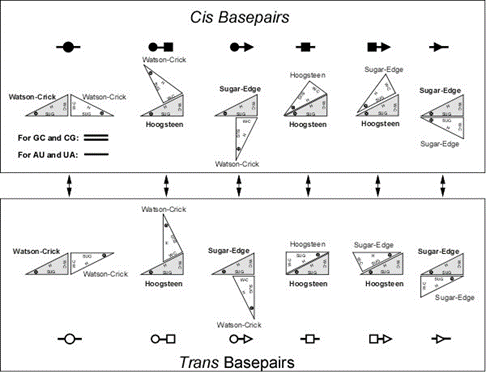
L’annotation Leontis-Westhof :
Géométries des interactions entre les nucléotides :
Il est bien
connu que les paires de bases canoniques sont des WC-WC Cis mais à
partir de là tout peut exister en termes d’interactions dans les
structures d’ARN.
La classification de Leontis-Westhof a
largement été adoptée par la communauté pour la
description d’interactions tridimensionnelles.
En vue de la complexité de ces
interactions une représentation schématisée a
été adopté. Cette schématisation permet
d’avoir une image de la conformation structurelle (d’identifier) “l’état
de la base”. Pour les paires de bases canoniques Watson Crick, représentée
avec un cercle, Hoogsteen edge représentée en carré et
Sugar edge ou le bord (coté) du sucre avec un triangle. Ces interactions
peuvent être de conformations Cis ou Trans (Figure 5A) elles sont représentées graphiquement en
plein noir et vide blanc respectivement. Un bord donné d'une base peut
potentiellement interagir sur un plan avec l'un des trois bords d'une seconde
base, et dans les conformations cis ou trans des liaisons glycosidiques (Figure
5B). ( Leontis
et Westhof,. 2001)
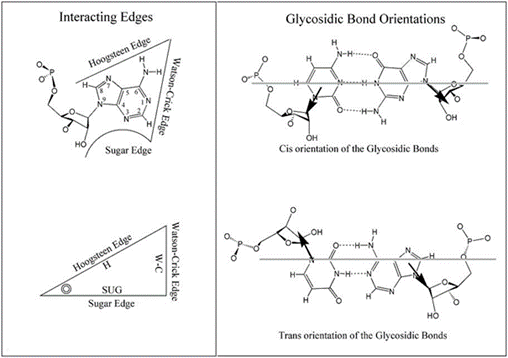 A
A
 B
B
Figure 5 : Géométrie des
interactions des bases nucléotidiques (Leontis & Westhof, 2001)
Motif structurel local kink turns
Les kink-turn sont extrêmement répandus dans les structures
d'ARN, avec des exemples dans la plupart des classes d'ARN fonctionnels. Il
existe de nombreux kink-turn différents dans l'ARN ribosomal des
bactéries, des archées et des eucaryotes. Le kink-turn ribosomal
Kt-7 de l'archéon Haloarcula
marismortui a été particulièrement bien
étudié (Figure 6).
Les motifs
Kink-turn sont des boucles internes récurrentes qui produisent des
virages nets (Kink en anglais) dans les hélices d’ARN (Lescoute et al., 2005).
On observe (Figure 6) onze sous structures d’ARN complètement
différentes (représentés en code couleur) mais elles ont
un sous morceau (motif structurel) similaire, une sous partie qui est
conservée entre les onze sous structures.

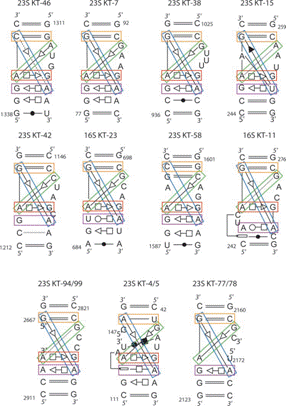
Figure 6 : 6-A
représentations du Backbone de neuf structures Kink-turn après superposition sur KT-7 6-B structures secondaire des motifs kink-turn (Klein et.al. 2001), (Lescoute et al., 2005)Les ARNs en
graphes (représentation) :
Une représentation graphique n’est
jamais complètement précise, car il est très difficile de
mettre en plan de ce qu’est la réalité, les ARNs
n’échappe pas à cela (Figure
7) est une représentation graphique d’une structure
d’ARN.
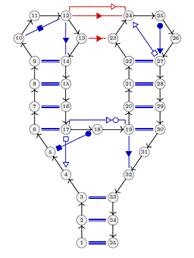
Figure 7 : représentation graphique d’ARN
Tout ce qui n’est pas des interactions
canoniques AU et CG ainsi que les G U paire de base oscillantes (wobble base
pairs) est considérés comme étant des interactions
tertiaires parce qu’ils nous donnent des informations sur la structure
tridimensionnelles (interactions et dispositions des nucléotides).
En observant cette représentation, une
question est soulevée : Existe-t-il des sous structures
conservées entre deux représentations graphiques d’ARN ?
Isomorphismes des sous graphes :
Il n’est
pas facile d’extraire de l’information des interactions
boucle-boucle, donc pour essayer de répondre à la question
posée, tout d’abord il est important de rappeler que :
➔
C’est
un problème d’une complexité NP-Difficile.
➔
Peut avoir un nombre exponentiel de résultats
(autant qu’il en existe de nucléotides).
Ensuite il est
nécessaire de formuler les hypothèses.
En se basant
sur des approches heuristiques tels que la méthode « branch and
cut » pour avoir toutes les solutions exhaustives :
1. Réduire les problèmes en
“tâches gérables”, décomposition des boucles.
2. Enlever les parties non nécessaire
(celles non impliquées dans des interactions).
3. Il est intéressant de prendre en
considération les connaissances biologiques préexistantes cela
permet d’introduire des restrictions au programme et donc de mieux
orienter l’étude :
●
Paires de boucles avec
interactions entre elles.
● Les nœuds doivent avoir au moins une interaction
Une similarité de sous graphes est
aussi appelé l’isomorphisme de sous graphes.
Les
étapes à suivre :
➢
Extension du sous-isomorphisme : Comparer des petits
morceaux
➢
Extensions de l’extension : Matcher les
“edges” et “arrêtes” là où ils
fusionnent, ils sont représentés sur un graphe appelé
“extension admissible”.
➢
Énumérer les solutions :
répéter jusqu’à identifier un graphe d'extension
admissible conservé donc une structure conservée.

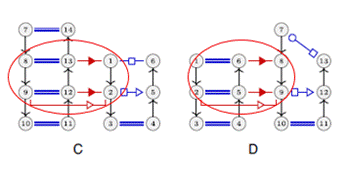
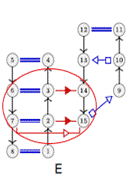
Figure 8 : la sous
structure A est retrouvé dans les structures C, D et E (Reinharz et al., 2018)
Carnaval
Le projet de Vladimir REINHARZ et ses collègues à amener
à une méthode entièrement automatisée d'extraction
et classification des sous-structures d'ARN, en fonction de leurs interactions
(les réseaux d’interactions récurrent ou RIN), les
séquences où le contexte n’est pas pris en
considération.
Les résultats de cette méthode
sont catalogués dans une base de données appelée CARNAVAL
décrit comme “Organized
collection of Recurrent Interaction Networks (RIN) in all experimentally
determined RNA structures”(http://carnaval.lri.fr).
Il est possible d’identifier les RIN en
entrant l’ID (identifiant) PDB (Protein Data Bank) de la structure ou
téléchargeant le fichier en format « cif »
(crystallographic information file) de la molécule. Cependant
après avoir essayé plusieurs dizaines de structures et
d’identifiant d’ARN aucune d’elles n’a donné un
résultat positif, certainement que les structures étant
récente ne sont pas encore introduite dans la base de données.
La base de données présente
également une classification des différents modules et motifs
avec recensement de leurs présences dans les structures qui existent :
❖
Le nombre de fois qu’elles sont observées.
❖
Et le nombre des interactions et avec quelles autres
RIN
Motif A-mineur
A mineur c’est deux paires de bases canoniques superposables qui
interagissent avec deux nucléotides qui sont souvent des « A
» d’où le nom A-mineur ; et est primordiale pour la
prédiction des structures 3D dont la distance a un rôle majeur.
Ce module structurel dépend seulement de la
géométrie elle-même ce qui le rend difficile à
manipuler car l’interaction entre les bases canoniques superposables et
les paires de bases se fait aléatoirement.
Il en existe deux types :
A. A-mineur de
type I : motifs répandus et important.
B.
A-mineur de type II (Figure 8 A) : est le produit d’un processus qu’on
appelle “L'HÉRITAGE”, il est l’adjonction entre l’A-mineur
de type I et le Ribose Zipper, le type II devient une boucle GNRA grâce
à d’autres interactions et puis une boucle A-rich (Reinharz et al,. 2018)
Traduction ribosomale
Lors de la traduction ribosomale,
l’ARNm possède un codon (1,2,3), l’ARNt possède un
anticodon (34,35,36) et le 16S ribosomal.
Pour que notre machine reconnaisse l’interaction codon-anticodon
il faut que les 3 paires canoniques se fasse et les 2 paires non canoniques
entre dans l’ARN messager qui va aider l’ARN de transfert avec le
16s (ensemble c’est le motif A mineur), cependant sans la présence
du potassium, le ribosome sera incapable de capter l’ARNt. (Figure 9)
La position 34 de l’anticodon et la 3eme position du codon sont
souvent modifiées chimiquement. Ces modifications chimiques sont
nécessaires pour que ces paires de bases puissent rentrer et avoir la
meilleure position pour que le A-mineur fonctionne et la où se repose la
difficulté de la régulation géométrique des
codon-anticodon.
(Les modification chimiques ne sont pas organisées mais ils sont
toujours la) (Figure 9)
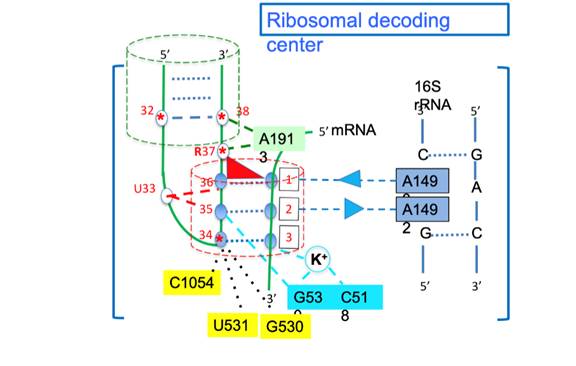
Figure 9 :
Traduction ribosomaleÉpigénétique
des ARNs et les modifications chimiques
Les modifications chimiques sont très importantes dans le monde
des ARNs et ce n’est pas que la méthylation de l’ADN qui
régule les gènes. Chez les ARNt, ARNm est absolument
nécessaire pour plusieurs raisons biochimiques, elles peuvent booster la
production de gènes ou la déstabiliser.
On a peu d’information sur ou est ce qu’elles se trouvent
dans la structure ? c’est quoi leur structure en générale ?
Et quel est ‘impact de ces modifications chimiques ?
Pour répondre
à ces questions des observations et des analyses ont été
faites et présenté pendant le séminaire et les
étapes d’études sont les suivantes :
1. Téléchargement
de tous les 150 milles structures 3D qui existe dans la PDB
2. Identification
des chaînes d’ARN qui possèdent au moins une modification
chimique
3. L’analyse
:
a. La distribution
des modification chimiques (Calcule des modifications chimiques pour seulement
les ARNs qui ont plus de 30 nt de toute la pdb)
b. La distribution
des types d’ARNs avec modifications chimiques
c. Distribution
nb. Nucléotides "proches" des nucléotides dans le
même ARN modifié
d. Distribution
nb. Nucléotides "proches" des nucléotides dans un autre
ARN
e. Distribution
nb. Nucléotides "proches" des protéines dans la
même protéine modifiée
Conclusion
La richesse de la combinatoire des paires de bases de
nucléotides permet aux molécules d'ARN de s'assembler en
réseaux d'interaction sophistiqués, qui sont utilisés pour
créer des sous-structures 3D complexes. L’approche
présentée sur les données pour extraire automatiquement
à partir de grands ensembles de données de structures 3D à
ARN complet les réseaux d'interactions récurrentes (RIN) nous permet
pour la première fois à détecter les réseaux
d'interaction reliant des composants distincts de la structure de l'ARN,
mettant en évidence leur diversité et leur conservation
grâce à des ARN fonctionnels non apparentés. Ainsi que le
modèle graphique qui permet d’effectuer des comparaisons par
paires de toutes les structures d'ARN disponibles et pour extraire les RIN et
les modules. Vladimir REINHARZ et
ses collègues ont rassemblé les résultats dans une base de
données en ligne (http://carnaval.lri.fr) qui seront
régulièrement mis à jour. (Reinharz et al.,2018)
Références
Djelloul, M. (2009). Algorithmes de graphes pour la recherche de
motifs récurrents dans les structures tertiaires d'ARN (Doctoral
dissertation).
Feng, L. (2012). Algorithmes pour l'étude de la
structure secondaire des ARN et l'alignement de séquences (Doctoral
dissertation).
Hendrix, D. K.,
Brenner, S. E., & Holbrook, S. R. (2005). RNA structural motifs: building blocks of a modular
biomolecule. Quarterly reviews of
biophysics, 38(3), 221-243.
Jean-Pierre, S.(2017). Exploration des
structures secondaires de l’ARN.
Klein, D. J., Schmeing, T. M., Moore, P. B., & Steitz,
T. A. (2001). The kink‐turn: a new RNA secondary structure motif. The EMBO journal, 20(15), 4214-4221.
Leontis, N. B., & Westhof, E. (2001). Geometric
nomenclature and classification of RNA base pairs. Rna, 7(4), 499-512.
Leontis, N. B., Lescoute, A., & Westhof, E. (2006). The
building blocks and motifs of RNA architecture. Current opinion in structural biology, 16(3), 279-287.
Lescoute, A., Leontis, N. B., Massire, C., & Westhof,
E. (2005). Recurrent structural RNA motifs, isostericity matrices and sequence
alignments. Nucleic acids research, 33(8), 2395-2409.
Mariette, J. (2017). Apprentissage statistique pour
l'intégration de données omiques (Doctoral dissertation).
Reinharz, V., Soulé, A., Westhof, E.,
Waldispühl, J., & Denise, A. (2018). Mining for recurrent long-range
interactions in RNA structures reveals embedded hierarchies in network
families. Nucleic acids research, 46(8),
3841-3851.
ROBERT, J. (1997). Etudes poursuivies.