Rapport de conférence de Monsieur Abdoulaye
Baniré Diallo
L’IA et la Bio-informatique au service de
l’agriculture de précision
Fait
par : Douja Meftah et Latifa Mohammadi

INTRODUCTION
AI, DONNÉES MULTI-OMICS QUELQUES
CONCEPTS ET DÉFINITIONS
INTELLIGENCE ARTIFICIELLE
DONNÉES MULTI-OMICS
PROBLÉMATIQUE
DE L’OPTIMISATION DE LA PRODUCTION LAITIÈRE
MÉTHODE ET ESSAI
COMPARAISON DES MODÈLES ET
RÉSULTAS
QUELQUES RÉSULTATS
DISCUSSION
CONCLUSION ET PERSPECTIVES
BIBLIOGRAPHIE
INTRODUCTION
Une richesse sans précédent de données biologiques a été générée par le
projet de séquençage du génome humain et des autres organismes, créant l'énorme
demande d'analyse et d'interprétation de ces données qui est gérée par
l'évolution de la science de la bioinformatique. Cette dernière est définie
comme l'application d'outils de calcul et d'analyse à la capture et à
l'interprétation de données biologiques [1]. Quant à l’intelligence artificielle
(AI) est une science dans laquelle les modèles mathématiques et
l'informatique sont omniprésents. Dans le cas de la biologie, l'IA s'appuie
largement sur les ressources et les techniques de la bioinformatique. La plupart des exemples d'IA qui font les gros titres de nos jours
(des ordinateurs jouant aux échecs aux voitures autonomes) reposent fortement
sur le Deep Learning (DL) et le traitement du langage naturel touchant particulièrement la robotique,
la communication, le traitement de texte et surtout dans la
reconnaissance et l'analyse d'images et de la voix (exemple, Google Duplex) [2].
Dans l’agriculture et surtout la production laitière est un secteur
agricole de base qui a fait l'objet d'innovation grâce à des méthodes basées
sur les données [3,4]. En fait,
l'augmentation de la collecte de données ouvre la porte aux approches du Deep
Learning la rendant indispensable pour les analyser et aider les agriculteurs à
prendre les bonnes décisions [4, 5].
Ce rapport fait à la suite de la présentation
Abdoulaye Baniré Diallo, sur L’intelligence artificielle et la Bioinformatique
au service de l’agriculture de précision, dans le cadre du cours BIO7002 du
programme de DESS en Bioinformatique à l'Université du Québec à Montréal. Dans
la première partie du rapport, les notions de base de l’intelligence
artificielle et des données Omics seront rappelées, puis à la deuxième partie,
la présentation de la méthode et des résultats en faisant une comparaison des
modèles seront présentés.
AI, DONNÉES MULTI-OMICS QUELQUES
CONCEPTS ET DÉFINITIONS
INTELLIGENCE ARTIFICIELLE
Les systèmes l'intelligence artificielle
(artificial intelligence, AI) intègrent souvent l'apprentissage automatique
(machine learning, ML) et l'apprentissage en profondeur (deep learning, DL)
pour créer une machine d'intelligence sophistiquée qui remplira bien les
fonctions humaines données. De plus en plus, les trois unités sont des pièces
individuelles de l'intégralité du puzzle du système d'intelligence artificielle
[6].
Dans la
science des big data, les méthodes d'apprentissage automatique sont des algorithmes
informatiques qui peuvent automatiquement apprendre à reconnaître des modèles
complexes basés sur des données empiriques [8, 9]. Le but d'une méthode
d'apprentissage automatique est de permettre à un algorithme d'apprendre des
données du passé ou du présent et d'utiliser ces connaissances pour faire des
prédictions ou des décisions pour des événements futurs inconnus [8, 9]. De
manière générale, le processus d'une méthode d'apprentissage automatique se
compose de trois phases :
1-
Construire le modèle à partir d'exemples d'entrées,
2-
Évaluer et régler le modèle,
3-
Mettre le modèle en production dans l'élaboration de
prévisions.
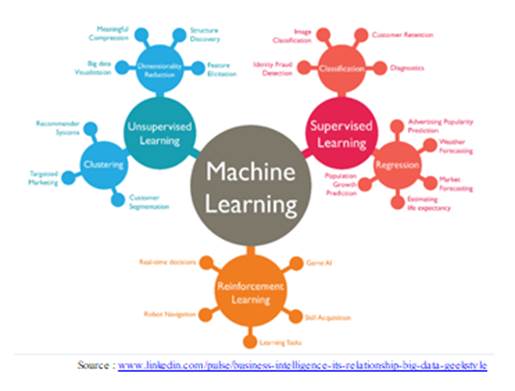
Figure 1- Types d'algorithmes d'apprentissage automatique
Les trois
paradigmes de base de l'apprentissage machine :
1- Apprentissage supervisé ("supervised learning")
L'apprentissage supervisé est la branche de
l'apprentissage automatique qui consiste à apprendre une fonction qui
cartographie une entrée à une sortie sur la base d'exemples de paires entrée-sortie
[10]. Il déduit une fonction a partir d’un ensemble de données d'entrainement
étiquetées (set of training examples) [11].
a. La
régression : L'analyse
de régression est largement utilisée pour la prédiction et la prévision, où son
utilisation chevauche considérablement le domaine de l'apprentissage
automatique.
Dans la modélisation statistique, l'analyse de régression est un
ensemble de processus statistiques permettant d'estimer les relations entre une
variable dépendante (souvent appelée « variable de résultat ») et une ou
plusieurs variables indépendantes. Cette méthode est principalement utilisée
pour prévoir et découvrir la relation de cause à effet entre les variables. Les
techniques de régression diffèrent principalement en fonction du nombre de
variables indépendantes et du type de relation entre les variables
indépendantes et dépendantes. [12, 13, 14].
b. Classification :
La classification est un exemple de la
reconnaissance de formes. Puisqu’elle permet d'identifier à quel ensemble de
catégories (sous-populations) appartient une nouvelle observation, sur la base
d'un ensemble d'apprentissage de données contenant des observations (ou
instances) dont l'appartenance à une catégorie est connue [15].
2- Apprentissage non supervisé ("unsupervised learning")
L'apprentissage non supervisé est un type
d'apprentissage hébbien auto-organisé qui permet de trouver des modèles
inconnus dans un ensemble de données sans étiquettes préexistantes. Il est
également connu sous le nom d'auto-organisation et permet la modélisation des
densités de probabilité des entrées données [16].
3-
Apprentissage par
renforcement ("Reinforcement learning")
A l’inverse de l'apprentissage supervisé apprentissage
par renforcement ne nécessite pas de présenter entrées / sorties étiquetées et
il n'est pas nécessaire de corriger explicitement les actions sous-optimales.
Au lieu de cela, l'accent est mis sur la recherche d'un équilibre entre
l'exploration (d'un territoire inexploré) et l'exploitation (des connaissances
actuelles) [17].
DONNÉES MULTI-OMICS
Les avancés
des technologies de séquençage à haut débit (ou séquençage de nouvelle
génération - NGS) ont permis l’émergence de nouvelles disciplines d'étude des
systèmes biologiques et ce dans leur intégrité, pavant la voie à la fondation
des sciences dites « omic » [18].
En effet, la multi-omic intègre diverses sources de données durant
l'analyse, par exemple l’analyse intégré génomique et transcriptomique.
Elles
dérivent leur nom de la source des données générées et incluent la
protéomique, la transcriptomique, la génomique, la métabolomique, la
lipidomique et l'épigénomique, qui correspondent aux analyses globales des
protéines, de l'ARN, des gènes, des métabolites, des lipides et de l'ADN méthylé
respectivement [19, 20, 21].
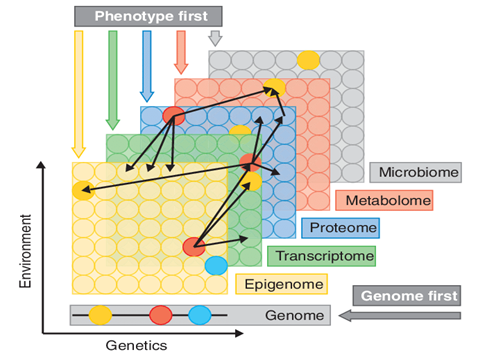
Figure 2- Types de données omiques
multiples et approches de la recherche sur les maladies [22]
Cette façon
de faire, i-e étude dans leur intégralité de ces différentes sources de données
afin de trouver une relation ou une association génotype-phénotype-environnement
cohérente, identifier des biomarqueurs pertinents et construire des modèles
élaborés reflétant un état physiologie normal ou pathologique [23].
Cette
intégration multi-omic est une avancée majeure dans le domaine scientifique et
n’aurai pas pu être réalisé sans la collaboration de plusieurs équipes
multidisciplinaires pour le développement des outils statistiques,
mathématiques, bioinformatiques et des stratégies expérimentales. L’intégration
et l’analyse de données à grande échelle nous a permis de passer d'une science
axée sur les modèles à une science fondée sur les données [24]. Effectivement,
l'intégration de l'analyse de données multi-omiques et de l'apprentissage
automatique a permis d'élaborer de nouvelles questions et d'identifier des
mécanismes biologiques dont on ne suspectait pas l’existence. L'identification de nouveaux biomarqueurs,
potentiellement exploitable cliniquement, devrait permettre le diagnostic plus
précis de maladies, une thérapie mieux adaptée aux besoins du patient et une
surveillance plus étroite de son évolution pavant la voie à la médecine
personnalisée [25, 26, 27].
Le service
OmicTools, une « metadatabase » manuellement curées en plus de
permettre l’accès à plus de 4400 outils accessibles sur le Web et liés à
l'analyse de données multi‑omique, ainsi que plus de 99 bases de données
sur le sujet [28, 29] est la résultante d’un besoin de faciliter l’accès aux
outils en la centralisant.
PROBLÉMATIQUE
DE L’OPTIMISATION DE LA PRODUCTION LAITIÈRE
La production
laitière représente un important marché en agriculture, avec un cheptel de près
de 1,4 million de vaches en 2014 qui génère des revenus nets de
6,17 milliards de dollars chaque année. Le Québec est la province qui
produit le plus grand volume de lait et possède le plus grand nombre
d'exploitations impliquées dans la production de lait [30, 31].
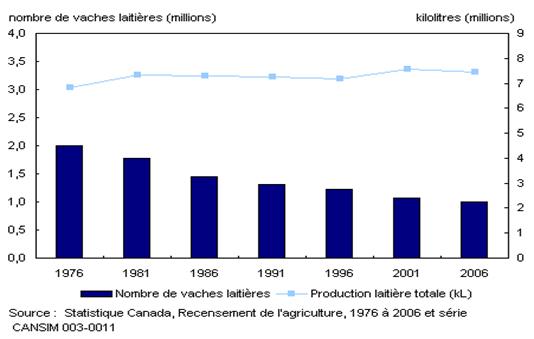
Figure
3- Évolution au cours des années de la production de lait
Plusieurs facteurs, associés
à diverses caractéristiques, ont une incidence sur les bénéfices de l'industrie
de la production laitière (figure 3). Les données de ces facteurs sont
collectées à partir de capteurs connectés, de tableaux de bord interactifs et
de questionnaires. Les facteurs sont interconnectés: par exemple,
l'environnement (comme la nutrition) peut avoir un impact direct sur la
production, mais il peut également tirer parti de la santé, de la génétique ou
de la gestion d'une vache, influençant ainsi la production. [32]
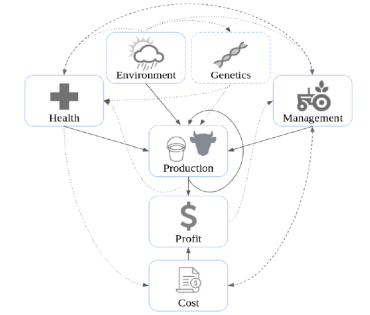
Figure 4- Diagramme de
relation des différents facteurs associés à l’industrie de la production de
lait [32]
Lors du séminaire,
M. Diallo a parlé de deux approches pour résoudre cette problématique :
Approche par ontologie : Les données sont organisées dans un
triplestore, sous format RDF et une ontologie OWL a été développé pour
faciliter la fédération des diverses sources concernant les divers aspects de
la production laitière (génotypes et phénotypes des animaux, pédigrée, etc.) puis
une fouille de caractéristiques typiques des vaches, sous la forme de graphes
de données répétitifs. Ces graphes devraient permettre d’alimenter les modèles
prédictifs par de features qui sont en même temps sémantiquement riches et
interprétables par l’utilisateur final, qu’il soit fermier ou agronome, ou
encore un spécialiste en qualité de lait. En outre, ils doivent faciliter la
comparaison entre animaux, troupeaux ou fermes à l’aide de mesures de
similarité.
Approche par données relationnelles (séries
chronologiques) : Dans
notre rapport, on va se concentrer sur cette approche sachant que les données
temporelles de la production laitière peuvent être définies comme un problème
de prédiction de séries chronologiques à partir de données multi-sources
(facteurs) et hétérogènes. [33]. Ainsi, les prévisions chronologiques peuvent
être utilisées pour analyser les séquences afin de détecter les tendances et
d'identifier les modèles, ce qui aide à construire un modèle prédictif précis.
MÉTHODE ET ESSAI
Nature
des données
Les données brutes proviennent de
25 années de tests faits sur 1,48M de vache dans 6670 troupeaux fournit par
Valacta et SAS.
|
Animal
|
Environnement
|
Production
|
|
Génétique
|
Troupeau
|
Analyses et tests
|
|
Pédigree
|
Nutrition
|
Prix de vente
|
|
Tempérament
|
Méteo
|
Tendances du marché
|
|
Santé
|
Géographie
|
Tableau I – Description des données utilisées
Des données son identifier par la date du
test et le numéro d’identité de chaque animal.
Et comprennent des mesures de divers composants : la production de
lait, le rendement en matières grasses, en protéines, en lactose; gestion de la
vache; dossiers de santé, etc. [32].
Cycle de lactation chez la vache
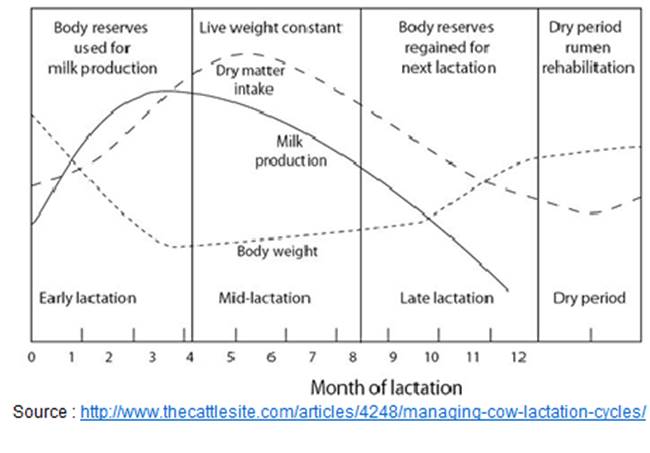
Figure 5- Cycle de lactation chez la vache Friesian : avec une représentation de la prise alimentaire, la production de lait
et le poids ce cycle est divisé en quatre phases, la lactation précoce, moyenne
et tardive (4 mois) et la période sèche (2 mois) avec de légères variations
possibles entre les individus selon les facteurs variable (race/génétique,
environnement, changement de saisons, nutrition; etc.).
Ré-indexation temporelle
La nature cyclique des données disponible
pour réaliser cette étude a facilité le choix de la technique de l’analyse des
séries chronologiques pour atteindre les objectifs de ce projet soit :
1- Identifier la nature des phénomènes
représenté par les séquences d'observations, découvrir les modèles cachés dans
les données.
2- Générer des prévisions des valeurs futures
de la variable de série chronologique et prédire le profit futur d’une vache déterminé.
Les séries chronologiques pour les vaches a
était alignées en utilisant indexée par l’identifiant unique de l’animal (ID)
et l'indice des mois après la naissance (MaB) [32].
Encoder
les variables catégorielles
L’encodage One-Hot était utilisé pour
convertir les variables non-ordinale sous une forme qui pourrait être fournie
aux algorithmes d’apprentissages machines. Toutes les fonctions ordinales sont
conservées telles quelles.
Le profit est calculé comme suit la valeur
quotidienne produite par une vache donnée a un âge précis moins son coût
journalier (alimentation ou santé) [32].
Imputé les valeurs manquantes
Avec la présence de valeurs manquantes dans
les ensembles de données, plusieurs types d'imputations sont effectués. La
moyenne mobile d’un intervalle de 3 a été utilisée pour les valeurs manquantes
des données de profit de fin de production.
Deux méthodes ont servi à imputer les
autres données manquantes :
1- Sans masquage : Remplissage des
valeurs manquantes restantes avec par une valeur de base donnée par expert;
2- Avec masquage : ignorer les horodatages t qui ont un profit
manquant. Remplissage des valeurs manquantes restantes[32].
Mise à
l’échelle des données (scaling)
Pour pouvoir comparer les différentes
caractéristiques entre elles une mise à l’échelle a été effectuer en utilisant
une fonctionnalité min max sur l’échelle [0,1]. Après toutes ces étapes, chaque
caractéristique était empilé dans un tenseur Ûi;t; f indexé par l’identifiant de animal
ID(i) et le MaB(t) les caractéristique f
. Les MaB était séparés en deux groupe
précoce (early test-dates) et tardif (late test-dates) qui vont correspondre
respectivement à l’entrée (input) Ûi;t early;f et la sortie output cible
p̃i, late dans le modèle[32].
La
sélection des caractéristiques
Les données les plus pertinentes pour cette
étude on était groupées par fonctionnalité et présenté dans le tableau 2. Feat : sont les fonctionnalités utilisées dans
analyse. Value : représente le minimum et le maximum de chaque fonction.
Les crochets sont des variables continues, les crochets doubles sont ordinaux,
Les accolades sont catégoriques. Legend :
est une brève description de la fonction
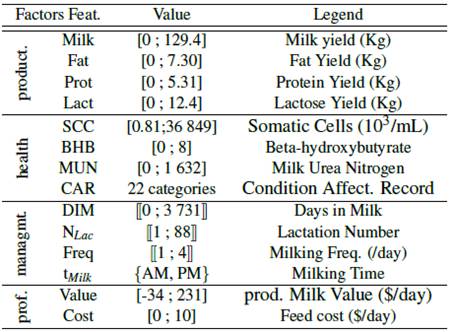
Tableau II - : Présentation de l'ensemble de données [32].
Entrée
/sortie
Le early test-date était définit à [18,46]
et le future test-date à [47;60] par un recoupage des données des vache qui ont
eu la première lactation entre 18 et 46 Mab et la deuxième entre 36 et 60 MaB
[32].
Sélection
des Vache
Pour la suite de l’étude uniquement les
vaches de race Holstein (92,8% dès vaches) a était garder. Toutes les vaches sans information sur la
valeur de leur lait, celle dont la valeur du lait était manquante depuis 6 mois
ou qui a été vendu entre MaB 18 et 60 ont était supprimés. Après toutes les
étapes de prétraitement, on se retrouve avec une fonction Ûi;t,f
contenant des informations continues sur 21 fonctionnalités, 417401 vaches
(28,2% restant) de MaB 18 à 60 [32].
Séparation
du set d’entrainement
Un jeu 33% des données des vache restantes à
était réserver (137742 vaches), Les modèles ont été entrainer avec les données
restante (279659 vaches) [32].
COMPARAISON DES MODÈLES ET RÉSULTAS
La base de la comparaison se fait selon les
trois mesures RMSE, MAE et biais.
La fonction objective est l'erreur
quadratique moyenne définie comme suit :
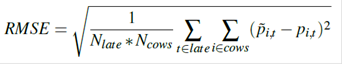
Nous utilisons également l'erreur absolue
moyenne:
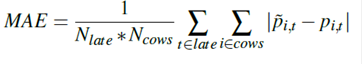
Nous utilisons également le biais:
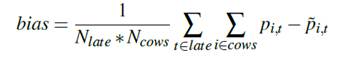
QUELQUES RÉSULTATS
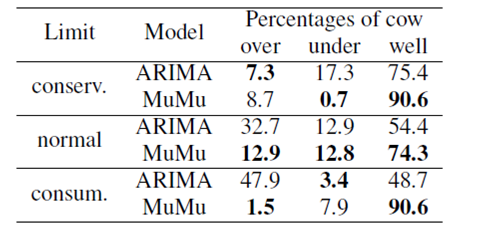
Tableau III: Erreurs de recommandation. Auto-ARIMA et MuMu sont comparés sur leurs
pourcentages d'erreurs de recommandation faites sur les vaches qu'ils ont
prédites [32].

Tableau IV: Loss test pour différents modèles. Auto-ARIMA réalise la pire prévision avec un RMSE de 14,04 $, MuMu
réalise le meilleur sans masquage: 8,36 $ [32].
|
|
UniMu-RNN
|
MuMu-RNN
|
Auto-Arima
|
Persistence Model
|
|
Présentation
|
Architecture faite de deux couches LSTM activées
à l'aide d'une tangente hyperbolique ;
Implémentation : la couche dense permet
d'éviter le surajustement et activer par une unité linéaire rectifiée [34].
Prédis le profit avec un réel
Utilise p̃i,early pour prédire p̃i,late
|
Utilise Ûi,early,f pour prédire p̃i,early ;
Même architecture et implémentation que UniMu ;
Fait entrer un tensor Ûi,early,f au lieu d'une matrice p̃i,early
|
Utilise pi;[18;46] pour prédire p̃i;[47;60].
Paramètre de stationnarité d [35].
Paramètre de saisonnalité D [36]..
Paramètres p, q, P et Q sont déterminés à
l'aide d'un algorithme pas à pas [37].
|
Modèle heuristique simple qui utilise la
valeur du profit réel précédent p̃i;t
comme prédiction p̃i,t+1
|
|
Implémentation
|
Formés sur les clusters informatiques :
Avec masquage : 2 Intel E5-2683 v4 Broadwell
CPUs (temps de formation: 4 jours)
Sans masquage : 4 x GPU NVIDIA
P100 Pascal (temps de formation: 13h).
Formation univariée nécessite 4 Go de
mémoire et multivariée 10 Go.
Utilise Adam Optimizer, une taille de lot
d'un (mettre à jour ses poids après chaque vache) Formé pour 30 époques en
utilisant le RMSE (avec Ncows = 1) = fonction objective
Keras 2.2.5 [38]. utilisé pour wrapper de
Tensorflow 1.13.1 [39].
|
Exécutés sur des ordinateurs locaux utilise
pyramid pour implémentation [40].
|
Exécutés sur des ordinateurs locaux
|
|
Évaluation
|
Évalués sur l'ensemble de test après
avoir été formé sur l'ensemble de formation à l'aide d'un ensemble de
validation de 20%.
|
Évalués sur l'ensemble des données car ils n'utilisent pas d'ensembles
d'entraînement.
|
|
|
MuMu est considéré comme le meilleur
modèle avec une RMSE plus petite (Table 2)
|
|
|
|
MuMu surpasse largement Auto-ARIMA avec
un biais très faible (-0,91% de p̃) et un meilleur RMSE et MAE, bien qu'il
ait toujours un RMSE relatif très élevé (62,2% de p̃) qui peut être considéré comme trop
grand pour être satisfaisant.
|
|
Tableau V : Résumé de la comparaison des modèles : UniMu, MuMu,
Auto-Arima et Persistence [32].
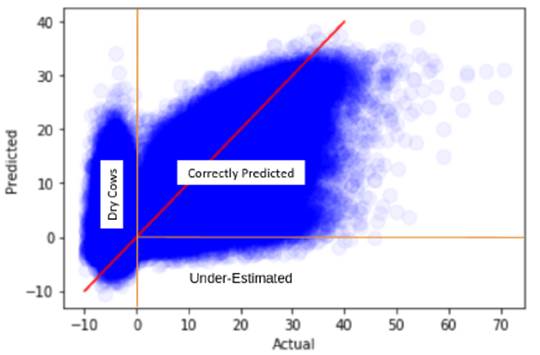
Figure
6 - Représente un plot des valeurs réelles (x) vs les valeur prédites (y) : du profit réalisé pour les vaches de 47 MaB du jeu de
données. On remarque l’existence de trois clusters le premier représentes les
vaches taries /période sèche (profit actuel négatif) et deuxième les vache sous-évalués
(le profil prédit est négatif) et le troisième les vaches correctement prédites
par le modèle [32].
On remarque que le profit réel générer par des vaches
taries est négatif puisqu’elles consomment sans générer de production alors que
le model prédit n’arrive pas à bien déterminer le profil réel des vaches et
sous-estime certaines d'entre elles.
DISCUSSION
Les données sont soumises avec beaucoup de
valeurs extrêmes dans le pipeline d'acquisition. Afin de réduire ce bruit, elles
doivent être corrigées et imputées pour ne pas biaiser la prédiction du modèle.
Malheureusement, un scaler Min-Max n'est pas robuste aux valeurs extrêmes ce
qui va empêchera un apprentissage efficace.
L'architecture pourrait être adaptée et
améliorée. Cependant, elle donne déjà de meilleurs résultats qu'une méthode
ARIMA à la pointe de la technologie. Les recherches futures mettront l'accent
sur le test d'architectures plus complexes, telles que la méthode d'ensemble
utilisant un autre type d'estimateur. Nous pourrions également implémenter une
architecture codeur-décodeur [41] ou un modèle récursif qui prédit pas à pas la
valeur suivante de chaque fonctionnalité.
Il a été bien mis en évidence que le modèle
ne parvient pas à prédire efficacement les vaches taries. Avoir un
classificateur prédisant l'état de l'animal (traite ou sec) avant le LSTM
pourrait être d'une grande aide pour prévenir ce biais et améliorer la
prédiction finale.
La santé et les marqueurs métaboliques tels
que le SCC, le MUN et le BHB peuvent aider à prédire la survenue de maladies. Dans
les recherches futures, il pourrait être utile de collecter des dossiers de
santé complets et de les inclure en tant que données d'entrée car, nous voyons
avec les codes CAR que différents types de conditions ont des influences
différentes sur la production de lait.
Le modèle typique de l'industrie laitière
ne convient qu'à une seule race. Le choix s’est porté sur les vaches Holstein
qui constituaient 92,8% de notre ensemble de données. Le modèle pourrait être
entrainé pour d'autres races et personnaliser pour chaque une d’elles.
CONCLUSION ET PERSPECTIVES
Les modèles présentés dans ce rapport illustrent
une première tentative de prédire le profit futur des vaches sur la base
d'informations précoces. Les modèles proposés obtiennent de meilleurs résultats
que le modèle statistique ARIMA malgré les
problèmes rencontrés, comme par exemple hétérogénéités les données, les
interactions complexes entre les variables et les données manquantes. Cela montre que les données peuvent être
utilisées pour améliorer les décisions prises par les agriculteurs et augmenter
leurs bénéfices.
BIBLIOGRAPHIE
1.
Bayat A. Science, médecine et avenir:
bioinformatique. (2002). BMJ . 324 (7344): 1018–1022. doi:
10.1136 / bmj.324.7344.1018
2.
Application de l'intelligence
artificielle et de l'apprentissage profond à quelques domaines de la biologie
site :http://biochimej.univ-angers.fr/Page2/COURS/Zsuite/6BiochMetabSUITE/5IntelligenceArtificielle/1IntelligenceArtificielle.htm
3.
Borchers M.,
Chang Y., Proudfoot K., Wadsworth B., Stone A., and Bewley J. (2017).
Machine-learning based calving prediction from activity, lying, and ruminating
behaviors in dairy cattle. Journal of dairy cattle. Journal of dairy science, 100(7):5664–5674.
4.
Ushikubo S., Kubota C., and Ohwada H. (2017). The
early detection of subclinical ketosis in dairy cows using machine learning
methods. In Proceedings of the 9th International Conference on Machine Learning
and Computing, pages 38–42. ACM.science, 100(7):5664–5674
5.
Kussul N.,
Lavreniuk M., Skakun S., and Shelestov A. (2017). Deep learning classification
of land cover and crop types using remote sensing data. IEEE
Geoscience and Remote Sensing Letters, 14(5):778–782.
6.
Kononenko I.
Machine learning for medical diagnosis: history, state of the art and
perspective. Artif Intell Med. 2001;23(1):89–109.
7.
Lane HY,
Tsai GE, Lin E. Assessing gene-gene interactions in pharmacogenomics. Mol Diagn
Ther. 2012;16(1):15–27.
8.
Landset S,
Khoshgoftaar TM, Richter AN, Hasanin T. A survey of open source tools for
machine learning with big data in the hadoop ecosystem. J Big Data.
2015;2:24.
9. Lin E, Tsai SJ. Machine learning and predictive algorithms for
personalized medicine: from physiology to treatment. In: Turnbull A, editor.
Personalized medicine. New York: Nova Science Publishers (in press).
10. Stuart J. Russell, Peter Norvig (2010) Artificial Intelligence: A Modern
Approach, Third Edition, Prentice Hall ISBN 9780136042594.
11. Mehryar Mohri, Afshin Rostamizadeh, Ameet Talwalkar (2012) Foundations
of Machine Learning, The MIT Press ISBN 9780262018258.
12.
Friedman J,
Hastie T, Tibshirani R. Regularization paths for generalized linear models via
coordinate descent. J Stat Softw. 2010;33(1):1–22.
13.
Zou H,
Hastie T. Regularization and variable selection via the elastic net. J R Stat Soc
Series B Stat Methodol. 2005;67(suppl):301–20.
14.
David A.
Freedman (27 April 2009). Statistical Models: Theory and Practice. Cambridge
University Press. ISBN 978-1-139-47731-4.
15.
Alpaydin,
Ethem (2010). Introduction to Machine Learning. MIT Press. p. 9. ISBN
978-0-262-01243-0.
16.
Hinton,
Geoffrey; Sejnowski, Terrence (1999). Unsupervised Learning: Foundations of
Neural Computation. MIT Press. ISBN 978-0262581684.
17.
Kaelbling, Leslie P.; Littman, Michael L.;
Moore, Andrew W. (1996). "Reinforcement Learning: A Survey". Journal of Artificial
Intelligence Research. 4: 237–
18.
Valeria
,D’Argenio (2018) “The High-Throughput Analyses Era: Are We Ready for the Data
Struggle?” High-Throughput,
7, 8; doi:10.3390/ht7010008
19.
Bersanelli,
Matteo; Mosca, Ettore; Remondini, Daniel; Giampieri, Enrico; Sala, Claudia;
Castellani, Gastone; Milanesi, Luciano (1 January 2016). "Methods for the
integration of multi-omics data: mathematical aspects". BMC
Bioinformatics. 17 (2): S15. doi:10.1186/s12859-015-0857-9. ISSN 1471-2105. PMC
4959355. PMID 26821531.
20.
Bock,
Christoph; Farlik, Matthias; Sheffield, Nathan C. (August 2016).
"Multi-Omics of Single Cells: Strategies and Applications". Trends in
Biotechnology. 34 (8): 605–608. doi:10.1016/j.tibtech.2016.04.004. PMC 4959511. PMID
27212022. Retrieved 31 October 2016.
21.
Vilanova
Cristina; Porcar Manuel (26 July 2016). "Are multi-omics enough?". Nature
Microbiology. 1 (8): 16101. doi:10.1038/nmicrobiol.2016.101. PMID 27573112.
22.
Hasin Y.,
Seldin M. & Lusis A. (2017). Multi-omics approaches to disease. Genome
Biol 18, 83 https://doi.org/10.1186/s13059-017-1215-1
23.
Tarazona, S., Balzano-Nogueira, L.,
& Conesa, A. (2018). Multiomics Data Integration in Time Series
Experiments. doi:10.1016/bs.coac.2018.06.005
24.
Schmidt, B.; Hildebrandt, A.
Next-generation sequencing: Big data meets high performance computing. Drug
Discov. Today 2017, 22, 712–717
25.
Garmire, Lana X.; Chaudhary,
Kumardeep; Huang, Sijia (2017). "More Is Better: Recent Progress in
Multi-Omics Data Integration Methods". Frontiers in Genetics. 8: 84.
doi:10.3389/fgene.2017.00084. ISSN 1664-8021. PMC 5472696. PMID 28670325.
26.
Tagkopoulos, Ilias; Kim, Minseung
(2018). "Data integration and predictive modeling methods for multi-omics
datasets". Molecular Omics. 14 (1): 8–25. doi:10.1039/C7MO00051K. PMID
29725673.
27.
Lin, Eugene; Lane, Hsien-Yuan
(2017-01-20). "Machine learning and systems genomics approaches for
multi-omics data". Biomarker Research. 5 (1): 2.
doi:10.1186/s40364-017-0082-y. ISSN 2050-7771. PMC 5251341. PMID 28127429.
28.
"Reap the rewards of a
biological insight engine". omicX. Retrieved 2019-06-26.
29.
Vincent J. Henry, Anita E. Bandrowski
Anne-Sophie Pepin, Bruno J. Gonzalez, and Arnaud Desfeux “OMICtools: an
informative directory for multi-omic data analysis”Database (Oxford). 2014;
2014: bau069. Published online 2014 Jul 14.
30.
Canadian Dairy Information Centre,
2015a. Dairy Animal Registrations. www.dairyinfo.gc.ca.
31.
Profile and impact of milk economy
site: http://lait.org/en/the-milk-economy/profile-and-impact-of-milk-economy/
32.
Gonçalves Frasco C, Radmacher M,
Lacroix R, Cue R, Valtchev P, Robert C, Boukadoum M, Sirard M-A and Diallo A B
(2020) Towards an Effective Decision-making System based on Cow Profitability
using Deep Learning. In Proceedings of 12th International Conference on Agents
and Artificial intelligence. ICAAT20-PP-247
33.
Box G. E., Jenkins G. M., and Reinsel
G. (1970). Time series analysis: forecasting and control holden-day san
francisco. BoxTime Series Analysis: Forecasting and Control Holden Day1970.
34.
Nair V. and Hinton G. E. (2010).
Rectified linear units improve restricted boltzmann machines. In Proceedings of
the 27th international conference on machine learning (ICML-10), pages 807–814.
35.
Chollet F. e. a. (2015). Keras. https://keras.io.
36.
Abadi M., Barham, P., Chen, J., Chen,
Z., Davis, A., Dean, J., Devin, M., Ghemawat, S., Irving, G., Isard, M., et al.
(2016). Tensorflow: A system for largescale machine learning. In 12th fusenixg
Symposium on Operating Systems Design and Implementation (fosdig 16), pages
265–283.
37.
Kwiatkowski D., Phillips P. C.,
Schmidt P., and Shin Y. (1992). Testing the null hypothesis of stationarity
against the alternative of a unit root: How sure are we that economic time
series have a unit root? Journal of econometrics, 54(1-3):159–178
38.
Hyndman R. and Khandakar Y. (2007).
Automatic time series forecasting: the forecast package for r 7, 2008. URL
http://www. jstatsoft. org/v27/i03
39.
Canova F. and Hansen B. E. (1995).
Are seasonal patterns constant over time? a test for seasonal stability.
Journal of Business & Economic Statistics, 13(3):237–252.
40.
Smith T. G. et al. (2017). pmdarima:
Arima estimators for Python. http://www.alkaline-ml.com/pmdarima.
41.
Cho K., Van Merri¨enboer B., Gulcehre
C., Bahdanau D., Bougares F., Schwenk H., and Bengio Y. (2014). Learning phrase
representations using rnn encoderdecoder for statistical machine translation.
arXiv preprint arXiv:1406.1078.









