Méthodes
de détection des transfert horizontaux de gènes
Rapport
sur la conférence présenté par Alix Boc
Par
Émilia Aïsha Coleman et Mélanie Dominique
BIF7002
- Séminaire de bioinformatique
Introduction
Le transfert de gènes horizontal (ou transfert latéral) est un mécanisme évolutif par laquelle une espèce a intégré un gène provenant d’une autre espèce dans son génome (Daubin et Szöllősi, 2016). Il a permis aux espèces de s’adapter à leur environnement au cours de l’évolution. Par exemple, il permet d’acquérir des facteurs de virulence et de la résistance aux antibiotiques chez les bactéries. De plus, il permet aussi de faire des réparations de gènes qui ont été endommagés. (Daubin et Abby ,2012)
z
Figure 1 : Trois types de transferts de gènes horizontaux (Furuya et Lowy, 2006)
Chez les procaryotes, les transferts horizontaux peuvent se faire de trois manières : par transformation, par conjugaison et par transduction (Figure 1). Lors de la transformation, le fragment d’ADN, dont l’origine est souvent d’organisme mort suite à une lyse ou une mort cellulaire, est absorbé dans le cytoplasme du procaryote à des fins nutritionnelles, de recyclage de nucléotide ou pour la sélection de gènes pour la recombinaison. Dans le cas de la transduction, le transfert se fait via un phage qui infecte son hôte afin de se répliquer jusqu’à la lyse de ce dernier. Ensuite, le phage récupère et intègre un fragment d’ADN de son hôte et le transporte jusqu’à un nouvel hôte. Dans certains cas, le phage injecte le fragment d’ADN qui a récupéré au lieu du sien. Certains procaryotes peuvent recruter des phages afin de mieux transférer leurs gènes. Enfin, la conjugaison est un mécanisme unilatéral qui se fait entre deux cellules. Le transfert de l’ADN se fait via un pilus sexuel qui lie le donneur (mâle) et le receveur (femelle). Le gène de conjugaison peut être transféré à l’aide des plasmides ou des phages de conjugaison, qui peuvent utiliser cette méthode afin de transférer le gène (Daubin et Szöllősi, 2016).
Pour les eucaryotes, c’est un peu plus complexe, car ces transferts doivent se faire très tôt dans leur développement cellulaire, c’est-à-dire au niveau des cellules germinales, ce qui est très difficile, car ces dernières sont isolées des cellules somatiques par des mécanismes qui empêchent les gènes étrangers de s’intégrer dans leur génome et de se transmettre à la progéniture. Chez les plantes, les TGH (transferts de gènes horizontaux) sont principalement présents dans le génome mitochondrial, qui contient souvent des gènes provenant de plantes différentes. Ces transferts se font via des parasites, des vecteurs tels que les insectes, des virus ou des agents pathogènes et par la pollinisation. Chez les animaux et les insectes, c’est surtout par une endosymbiose entre ceux-ci et des bactéries (Huang, 2013).
Alix Boc et Vladimir Makarenkov ont développé un algorithme de détection de transfert de gènes horizontal complet étant disponible sur T-Rex sous le nom de HGT-Detection. De plus, ce logiciel peut comparer des arbres par une nouvelle méthode appelée la dissimilarité de bipartitions, qui améliore la détection des THG. Ils ont validé les scénarios de transfert par bootstrap en comparant les scénarios des réplicats avec l’original. Ils ont aussi défini des règles d’évolution tels que l’interdiction des transferts croisés et dans la même lignée, ainsi que la contrainte des sous-arbres qui permet le transfert de sous-embranchement présent dans l’arbre d’espèces uniquement s’il est aussi présent dans l’arbre de gènes. Enfin, ils ont développé deux algorithmes de détection partiels afin de détecter les gènes mosaïques qui ont des sous-séquences d’origines différentes (Boc, 2018). Dans ce rapport, nous allons utiliser le logiciel HGT-Detection afin de détecter les transferts du gène de l’ultrabithoraxe (Ubx) dans le sous-embranchement des Hexapodes.
Méthodologie
Création d’un arbre de gènes et un arbre d’espèce
Pour faire la recherche, le logiciel Orthoscope (Inoue et Satoh, 2019) a été utilisé en premier. Il fallait d’abord déterminer quels groupes et quelles espèces étaient présents dans la base de données pour permettre la création de l’arbre de gènes. Quelques espèces différentes, plus ou moins similaires, ont été sélectionnées afin de voir les variations présentes selon le groupe. Pour ce rapport, le taxon des Hexapoda a été choisi. C’est un embranchement qui regroupe principalement des insectes, pour lesquels le transfert de gènes étudié était l’ultrabithoraxe, qui nécessaire à la régulation de la morphogénèse. Les séquences obtenues, en format FASTA, ont été soumises au site EBI (European Bioinformatics Institute) afin de créer un nouvel arbre phylogénique à partir de MAFFT (Multiple Sequence Alignment) (Madeira et al. 2019). Seulement 7 espèces différentes (Heliconis melpomene, Danaus plexippus, Bombyx mori, Bombus impatiens, Apis mellifera, Acyrthosiphn pisum et Drosphila melanogaster) ont été analysées puisque c’est le nombre maximum de séquences qu’il est possible de soumettre sur le site Orthoscope. L’utilisation de ce site est essentielle puisqu’il permet de fournir l’arbre de gènes qui sera utilisé pour déterminer le transfert de gènes par la suite à partir des séquences FASTA envoyées. Afin d’avoir une recherche plus ciblée sur le groupe taxonomique (et non sur l’ensemble des espèces présentent dans la base de données), il est possible de soumettre le nom des espèces à analyser dans un fichier à part en format texte. Ceci donnera un arbre de gènes plus court à analyser et plus facile à manipuler sur T-Rex.
Utilisation du logiciel HGT de T-Rex (Boc et al, 2010)
Il faut utiliser le format Newick obtenu à partir de l’alignement MAFFT créé pour ce qui est de l’arbre des espèces. Pour ce qui est de la séquence de gènes, c’est celle produite par Orthoscope. Puisque l’arbre des espèces est créé avec seulement 7 espèces, mais que l’arbre de gènes est créé avec un éventail plus grand, il faut s’assurer de remplacer les noms d’espèces appropriées avec les mêmes lettres. Par exemple l’arbre :
((A:1.0, E:1.0), B:1.0, (C:1.0, D:1.0):1.0);
contiendra plus de lettres avec Orthoscope puisque plus d’espèces sont prises en compte.
Tableau I : Liste des espèces avec leur lettre correspondante
|
Lettre associée |
Espèce |
|
A |
Heliconius melpomene |
|
B |
Danaus plexippus |
|
C |
Bombyx mori |
|
D |
Bombus impatiens |
|
E |
Apis mellifera |
|
F |
Acyrthosiphon pisum |
|
G |
Drosophila melanogaster |
Résultats

Figure 2 : Arbre phylogénétique (MAFFT)
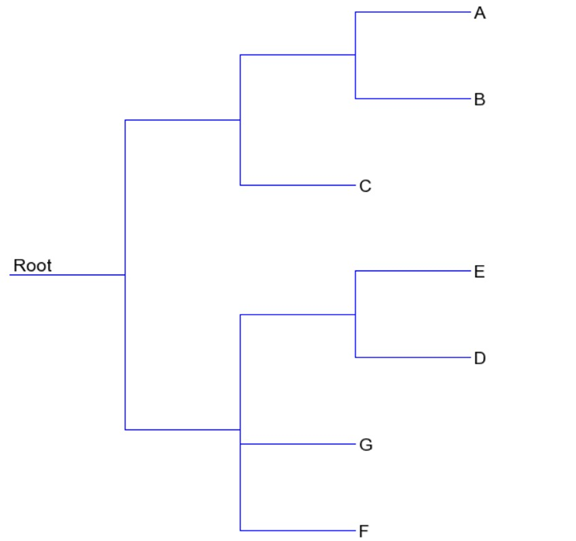
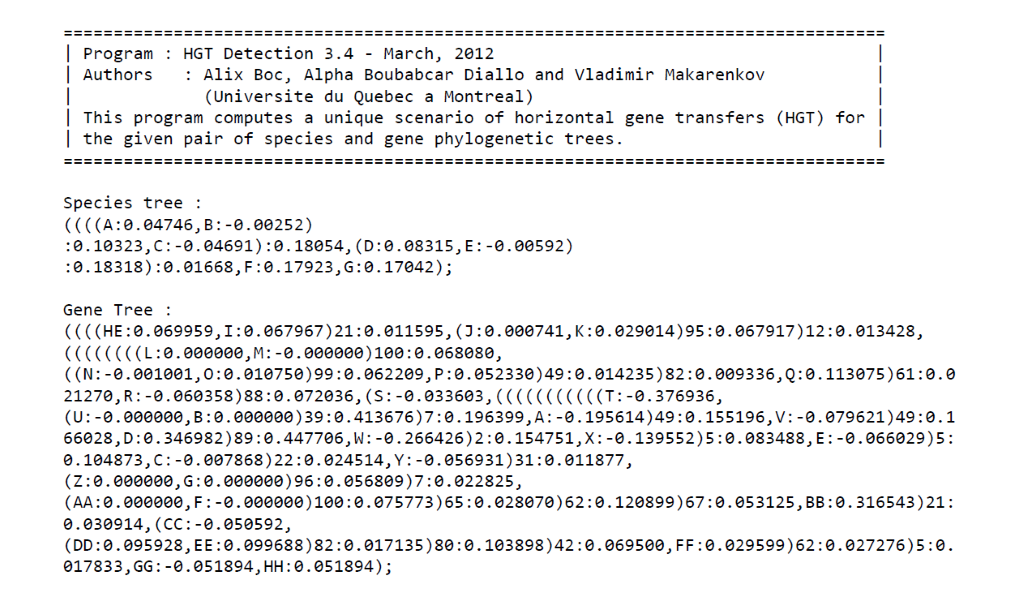
Figure 3 : Arbre d’espèces selon T-Rex
Avec les deux arbres d’espèces (Figures 2 et 3), il est possible d’observer que l’embranchement est presque identique pour les deux, sauf concernant la partie inférieure de l’arbre pour les espèces G et F (Acyrthosiphn pisum et Drosphila melanogaster respectivement).
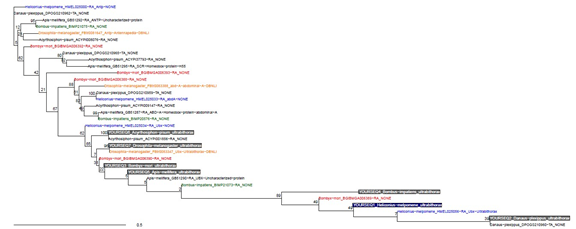
Figure 4 : Arbre phylogénétique obtenu sur Orthoscope
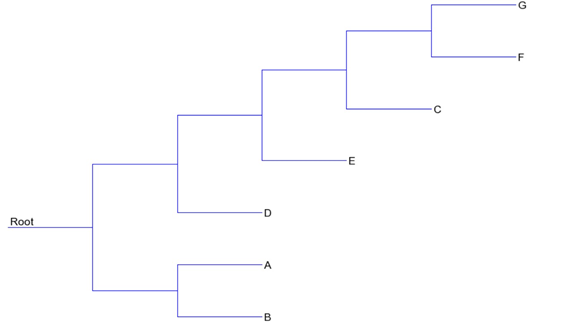
Figure 5 : Arbre de gènes selon T-Rex
Les deux arbres de gènes obtenus sont différents puisque sur Orthoscope, une plus grande liste d’espèces est prise en compte, alors que pour T-Rex, seulement les espèces d’intérêt sont comparées (Figure 4 et 5). On peut observer le même type d’arbre dans les deux cas, en s’attardant seulement sur les espèces d’intérêt (arbres inversés).
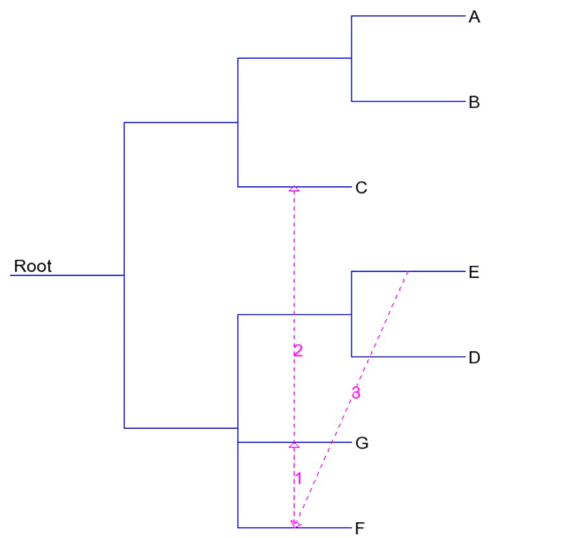
Figure 6 : Résultats de transfert de gènes
Pour ce qui est des résultats des transferts de gène (Figure 6), il y en a trois qui ont été notés. C’est-à-dire deux transferts normaux et un trivial (transfert de gènes dans le même nœud). Le transfert 1 est celui entre Acyrthosiphon pisum et Drosophila melanogaster. Le transfert 2 est le transfert de gènes du groupe Acyrthosiphon pisum et Drosophila melanogaster à l’espèce du groupe C, Bombyx mori. Pour le transfert 3, le gène de l’espèce E, Apis mellifera au groupe C, F, G, soit : Bombyx mori, Acyrthosiphon pisum et Drosophila melanogaster.
Discussion
Le logiciel HGT de T-Rex a permis de détecter trois transferts de gènes chez les espèces du taxon des Hexapodes. Cependant, pour avoir eu ces résultats il a dû éliminer plusieurs branches de l’arbre de gènes obtenu par Orthoscope afin de générer un arbre de gène qui présente mieux les espèces d’intérêt et qui est comparable à son arbre d’espèces. Il y a donc eu une perte d’informations qui auraient pu influencer les résultats, car plusieurs espèces ont été éliminées afin d’obtenir deux arbres ayant le même nombre de branches. Cette perte d’information aurait pu être problématique si on avait voulu utiliser ces arbres pour comparer des transferts de gènes différents entre d’autres espèces.
Le transfert de gène horizontal peut se faire à la fois dans le génome cœur (présent dans toutes les populations de sous-espèce) et le génome accessoire (présent dans une ou plusieurs sous-populations et qui peut être remplacé ou éliminé). Il est donc difficile de déterminer si les changements évolutifs détectés dans les arbres de phylogénie sont dus à un transfert horizontal ou à une perte de matériel génétique (Boto, 2010). Certains transferts horizontaux, par exemple les plus anciens ou les mieux intégrés, peuvent ne pas être détectés par les différentes approches méthodologiques. De plus, les transferts entre les feuilles sœurs peuvent être silencieux au niveau phylogénique (Ragan et al, 2009).
Selon Boto et al, certains chercheurs ont même suggéré que le transfert ne se fait pas ou très rarement au niveau des gènes cœurs, contrairement aux gènes accessoires. De plus, d’autres études ont aussi démontré que les transferts horizontaux ont aussi tendance à se faire entre les sous-espèces qui sont très proches dans l’arbre (ayant une distance relativement très faible), comparé aux espèces plus éloignées (Boto, 2010). Enfin, certains transferts peuvent ne pas bien s’intégrer dans le génome et ne persistent pas dans l’évolution. (Ragan et al, 2009).
Cela est surement dû à un certain mécanisme de préservation afin de conserver l’intégrité de l’espèce, par exemple, certains transferts de gène peuvent causer des problèmes en rendant la nouvelle souche stérile ou non viable, ce qui est souvent le cas chez les espèces non procaryotes. Dans le cas des gènes accessoires, puisque leur rôle est plus au niveau de la différenciation des sous-espèces, ils ont plus tendance à se transférer entre les sous-espèces afin de leur donner de nouvelles caractéristiques à des fins de survie ou de résistance (Raoult et Koonin,2012).
Dans le cas du gène Ubx, qui est grandement impliqué dans la morphogénèse chez les insectes, il est difficile de croire que celui-ci ferait partie du génome accessoire, car selon les analyses par Orthoscope, il présent chez plusieurs espèces d’insectes et n’est donc pas limité à des sous-populations d’espèces. Par contre, il y a grande possibilité que celui-ci peut se déplacer dans les différents génomes, tout en restant dans le génome cœur.
En effet, selon Peccoud et al (2017), la majorité des transferts de gènes horizontaux chez les eucaryotes sont des transferts d’éléments transposables du génome, c’est-à-dire des morceaux d’ADN pouvant se déplacer à différentes régions dans le génome. En analysant 195 espèces d’insectes regroupées en 81 lignées, ils ont découvert pas moins de 2248 transferts horizontaux d’éléments transposables du génome durant les dernières 10 millions d’années ce qui a grandement contribué à l’évolution des insectes. Sur les 195 espèces, 176 d’entre elles et donc 71 lignées sur 82 étaient impliquées dans au moins un transfert horizontal (Peccoud et al, 2017).
Conclusion
La perte de branches plus anciennes a pu éliminer la possibilité de détecter des transferts de gènes plus anciens et donc une possibilité de transfert de gènes faisant parties du génome cœur ayant mieux intégré dans le génome des espèces du sous-embranchement des Hexapodes. Malgré cela, les transferts ayant été détectés par T-Rex peuvent sous-entendre que des éléments transposables du gène Ubx c’est transféré dans les génomes des différentes espèces.
Références
Boc, A., (2018). Détection des transferts horizontaux de gènes, BIF7002 — Séminaire de bioinformatique, Université du Québec à Montréal.
Boc, A., Philippe, H. and Makarenkov, V. (2010), Inferring and validating horizontal gene transfer events using bipartition dissimilarity, Systematic Biology, 59: 195-211. (T-Rex)
Boto L. (2010). Horizontal gene transfer in evolution: facts and challenges. Proceedings. Biological sciences, 277(1683), 819–827. DOI : https://doi.org/10.1098/rspb.2009.1679
Daubin, V., Abby, S. (2012), Les transferts horizontaux de gènes et l’arbre de la vie, Med Sci (Paris) 28 (8-9) 695-698, DOI : https://doi.org/10.1051/medsci/2012288007
Daubin, V. Szöllősi, G. J. (2016). Horizontal Gene Transfer and the History of Life. Cold Spring Harbor perspectives in biology, 8(4), a018036. DOI : https://doi.org/10.1101/cshperspect.a018036
Furuya, E., Lowy, F., (2006). Antimicrobial-resistant bacteria in the community setting. Nat Rev Microbiol 4, 36–45. DOI : https://doi.org/10.1038/nrmicro1325
Huang J. (2013). Horizontal gene transfer in eukaryotes: the weak-link model. BioEssays : news and reviews in molecular, cellular and developmental biology, 35(10), 868–875. DOI : https://doi.org/10.1002/bies.201300007
Inoue, J., Satoh, N., (2019) ORTHOSCOPE : An Automatic Web Tool for Phylogenetically Inferring Bilaterian Orthogroups with User-Selected Taxa, Molecular Biology and Evolution, 36(3) 621–631, DOI: https://doi.org/10.1093/molbev/msy226 (ORTHOSCOPE)
Madeira F, Park YM, Lee J, et al. (2019), The EMBL-EBI search and sequence analysis tools APIs in 2019. Nucleic Acids Research. Jul; 47(W1) : W636-W641. DOI : http://doi.org/10.1093/nar/gkz268 (MAFFT)
Peccoud, J., Loiseau, V., Cordaux, R., Gilbert, C., (2017). Horizontal transfer of transposons in insects, Proceedings of the National Academy of Sciences May 2017, 114 (18) 4721-4726; DOI : https://doi.org/10.1073/pnas.1621178114
Ragan, M. A., & Beiko, R. G. (2009). Lateral genetic transfer : open issues. Philosophical transactions of the Royal Society of London. Series B, Biological sciences, 364(1527), 2241–2251. https://doi.org/10.1098/rstb.2009.0031
Raoult, D., Koonin, E. V. (2012), Microbial genomics challenge Darwin, Frontiers 2(116), 99. DOI : https://doi.org/10.3389/fcimb.2012.00127
Annexe
Séquences en format FASTA des espèces choisies
>Heliconius-melpomene_ultrabithorax
AGEQQYAKADCSKAGGEQQNGYGGKEAWGSGLGALVRPAACTPEARYSESSSPGRALPWGNQCGLPGAAASAQPVQHQPTNHTFYPWMAI
>Danaus-plexippus_ultrabithorax
MNSYFEQGGFYGAHGVHQGGGGGDQYRGFPLGLTYAQPHALHQPRPQDSPYDASVAAACKLYAGEQQYAKADCSKAGDQQNGYGGKEAWGSGLGALVRPAACTPEARYSESSSPSRALPWGNQCALPGAAASAQPVQHQPTNHTFYPWMAIAECELDTKEKIVSY
>Bombyx-mori_ultrabithorax
MNSYFEQGGFYGAHGVHQGGGGGDQYRGFPLGLTYAQPHALHQPRPQDSPYDASVAAACKLYAGEQQYPKADCSKPGGEQQNGYGGKEAWGSGLGALVRPAACTPEARYSESSSPGRALPWGNQCALPGSAASAAQPVHQQPTNHTFYPWMAIAGANGLRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHALCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAAAAAAAAAAAQGHPEH
>Bombus-impatiens_ultrabithorax
MNSYFEQTAGGFYGSHHHQTGAASQHHDPATAAAYRGFPLGLGMSPYASTQHHHHTSSSLGIHPGGGTNTRPPQDSPYDASVATACKLYSTTPEATGHTTSSYSTTATKDCKQQDQASAHQNGYAAVMAAAAVKDVWQSATSGSNSQSNSVVRPSACTPEGTRVGSYGGLVGGDPASSPGNNSSSRSLTSSWNTCSLNSSASQPVATQLHQQPTNHTFYPWMAIAGKEDIAKPHWTWLQGKRSARS
>Apis-mellifera_ultrabithorax
MNSYFEQTAGGFYGSHHHQTGAASQHHDPATAAAYRSFPLGLGMSPYASTQHHHHTSSSLGIHPGGGTNTRPPQDSPYDASVATACKLYSTTPEATGHTTSSYSTTAAKDCKQQDQASAHQNGYAAVMAAAAVKDVWQSATSGANSQSNSVVRPSACTPEGTRVGSYGGLVGGDPASSPGNNSSSRSLTSSWNTCSLNSSASQPVATQLHQQPSNHTFYPWMAIAGANGMRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHSLCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAAAAAAAAHQQQAAGGGPEGAN
>Acyrthosiphon-pisum_ultrabithorax
MNSYFEQSGFYGGHHHHQSSSAHHHDQNAVAAAYRFPIGLGMSPYASSQHHHHHGLSQSRAPQDSPYDASVAAAACKLYGSSGGGGGGGGVGGGGGGGGGGGSGGGGGDQSSAAAVAAAGLNYTKTEPCVGKSDAAAAAHQNGYASSPKDVVWPGNAASAAANGSLLRPNVCTPESRVGYGSGNSVSGLDGAGASPNSARNSAGSLSASWNTCTGINNNGHQQQALQLNQQQNNHTFYPWMAIAGQSLGANGLRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHALCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAVLAAAQAAGDR
>Drosophila-melanogaster_ultrabithorax
MNSYFEQASGFYGHPHQATGMAMGSGGHHDQTASAAAAAYRGFPLSLGMSPYANHHLQRTTQDSPYDASITAACNKIYGDGAGAYKQDCLNIKADAVNGYKDIWNTGGSNGGGGGGGGGGGGGAGGTGGAGNANGGNAANANGQNNPAGGMPVRPSACTPDSRVGGYLDTSGGSPVSHRGGSAGGNVSVSGGNGNAGGVQSGVGVAGAGTAWNANCTISGAAAQTAAASSLHQASNHTFYPWMAIAGECPEDPTKSKIRSDLTQYGGISTDMGKRYSESLAGSLLPDWLGTNGLRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHALCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAAAAAAAAAVQGGHLDQ
Séquences en format FASTA des espèces utilisées pour l’arbre de gènes par Orthoscope
>Heliconius-melpomene_HMEL025000-RA_Antp-NONE
MSANNCDSMTYFSNAYMPDMRNGGGHDHQQAHAHYGAVPQQGHEMEGCDQQLRPAQHHYPAQPAPGMPYPRFPPYDRLGYYQQMEQNGYRPDSPSQMGHMGPKTDGYGPNGHQPPTPAVYTSCKLQAAAATAGGVPGSPPLEQAQQMPHHMHPQQHMAQHGMPPHQQHLMYPVDDMQHQTQMPPMHQQSMHAQQAPPQQPPPNTNASLPSPLYPWMRSQFERKRGRQTYTRYQTLELEKEFHFNRYLTRRRRIEIAHALCLTERQIKIWFQNRRMKWKKENKTKGEPGSGDEPDNMSPPTSPQ
>Danaus-plexippus_DPOGS210962-TA_NONE
MRSQFERKRGRQTYTRYQTLELEKEFHFNRYLTRRRRIEIAHALCLTERQIKIWFQNRRMKWKKENKTKGEPGSGDEPDNMSPPTSPQ
>Bombyx-mori_BGIBMGA006392-RA_NONE
MQRCALRLLKLCDFVWAGQTNERKRGRQTYTRYQTLELEKEFHFNRYLTRRRRIEIAHALCLTERQIKIWFQNRRMKWKKENKTKGEPGSGDEPDNMSPPTSPQ
>Acyrthosiphon-pisum_ACYPI37793-RA_NONE
MNMSSYQFMNCYQQSQQAQQQQGRAVTSPVDPLQTGNSPGGGGAASASAADYYGQAAQGGGGGGGVYNAGPAAGCYSPQQYASQYMQQSSPSMIDYTQLHSVNHQQQHQQHQQHQRLQAAAAAAAAGAHLQHLQHHGGGGGVGGLPSPGGGGAISPISSALNNNVVGMPSAAGGVVQCKYEVTASASATPNGISSPQDLTTSAAPPPGSRSPPPAVVMKSVRSSGGPVAGSSPATQGPVSQTSSSPASSISSTSSNGGGGGGNGNGPASSGGKQGSGQNPPQIYPWMKRVHLGQSTVNANGETKRQRTSYTRYQTLELEKEFHFNRYLTRRRRIEIAHALCLTERQIKIWFQNRRMKWKKEHKMAQHEYGAVPLSHGRPALRHPVPLHAPHHLRFVLLI
>Apis-mellifera_GB51295-RA_SCR-Homeobox-protein-H55
MSSYQFVNSLASCYAGQQAQQQPRATANSPGEPLQAASPAGGDYYNPNAATTSYPAPCYSPQQHYPQHPYATPASGMQHTAPTGMIDYTQLQPQPRLTAASLHQQQQHHPQQQHHPQAQQQQQQQQQQLHQDPTAPLLQSGAPSAPSASSCKYADSTGSTGVASPQDLTTSTSRNSPTPLVAAAAAGTSKAVGTAGLTSPPGASSRSSAAAAAAASPASGSSRPQEGSSALTTASSASSPASSTSSTSSTGNNSSNKNNPTGNEPPKIYSWMKRVHIGQSTVNANGEVKRQRTSYTRYQTLELEKEFHFNRYLTRRRRIEIAHALCLTERQIKIWFQNRRMKWKKEHKMASMNIVPYHMSPYGHPYQFAPHPGQFAHLAT
>Danaus-plexippus_DPOGS210965-TA_NONE
MTGDSCGRQISPRAPTDYECTVNANGETKRQRTSYTRYQTLELEKEFHFNRYLTRRRRIEIAHALCLTERQIKIWFQNRRMKWKKEHKMASMNIVPYHMNPYGHPYQFDLHPSQFAHLSA
>Bombyx-mori_BGIBMGA006393-RA_NONE
MSSAATTNRVSDMWNNYVNTSDPNSKQSKVYNYKQYPTMANNYYNYQNYYPHNGCYGNVLSANCSEKLCGTPTEGIVKSEPNWHGYSANLQTGSNGMDAIKKWQEMNQYTQQQYGNYNYNQTNMLCNTSVVEKVTDVRSMNSPGQCSISENYGSPQSVTSDFKSISPLGDDSPHLRALLTKPKTKSQNQPYFPACDNSSTREISQQVPYVENNNWEKNNETTSGKECNLSQFHGGFQKVDDQTSIKKDAVGGAPSPNEVAQSSMGAAEPCQDTMTRVQAVGDNADYTENKMAAAPEVQAYYPWMKGVSGDTKKEGSKRTRQTYTRFQTLELEKEFHFNKYLSRRRRIEVSHALGLTERQIKIWFQNRRMKAKKDGKLTTSPEPFALDDIGATKLGNNVSEYIDPRQQIGLPEYPNYHAGSVQPNIGHIPENMSHCMMPPYGGMLPKM
>YOURSEQ6_Acyrthosiphon-pisum_ultrabithorax
MNSYFEQSGFYGGHHHHQSSSAHHHDQNAVAAAYRFPIGLGMSPYASSQHHHHHGLSQSRAPQDSPYDASVAAAACKLYGSSGGGGGGGGVGGGGGGGGGGGSGGGGGDQSSAAAVAAAGLNYTKTEPCVGKSDAAAAAHQNGYASSPKDVVWPGNAASAAANGSLLRPNVCTPESRVGYGSGNSVSGLDGAGASPNSARNSAGSLSASWNTCTGINNNGHQQQALQLNQQQNNHTFYPWMAIAGQSLGANGLRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHALCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAVLAAAQAAGDR
>Acyrthosiphon-pisum_ACYPI001856-RA_NONE
MNSYFEQSGFYGGHHHHQSSSAHHHDQNAVAAAYRFPIGLGMSPYASSQHHHHHGLSQSRAPQDSPYDASVAAAACKLYGSSGGGGGGGGVGGGGGGGGGGGSGGGGGDQSSAAAVAAAGLNYTKTEPCVGKSDAAAAAHQNGYASSPKDVVWPGNAASAAANGSLLRPNVCTPESRVGYGSGNSVSGLDGAGASPNSARNSAGSLSASWNTCTGINNNGHQQQALQLNQQQNNHTFYPWMAIAGQSLGANGLRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHALCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAVLAAAQAAGDR
>YOURSEQ7_Drosophila-melanogaster_ultrabithorax
MNSYFEQASGFYGHPHQATGMAMGSGGHHDQTASAAAAAYRGFPLSLGMSPYANHHLQRTTQDSPYDASITAACNKIYGDGAGAYKQDCLNIKADAVNGYKDIWNTGGSNGGGGGGGGGGGGGAGGTGGAGNANGGNAANANGQNNPAGGMPVRPSACTPDSRVGGYLDTSGGSPVSHRGGSAGGNVSVSGGNGNAGGVQSGVGVAGAGTAWNANCTISGAAAQTAAASSLHQASNHTFYPWMAIAGECPEDPTKSKIRSDLTQYGGISTDMGKRYSESLAGSLLPDWLGTNGLRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHALCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAAAAAAAAAVQGGHLDQ
>Drosophila-melanogaster_FBtr0083347_Ubx-Ultrabithorax-DBNLI
MNSYFEQASGFYGHPHQATGMAMGSGGHHDQTASAAAAAYRGFPLSLGMSPYANHHLQRTTQDSPYDASITAACNKIYGDGAGAYKQDCLNIKADAVNGYKDIWNTGGSNGGGGGGGGGGGGGAGGTGGAGNANGGNAANANGQNNPAGGMPVRPSACTPDSRVGGYLDTSGGSPVSHRGGSAGGNVSVSGGNGNAGGVQSGVGVAGAGTAWNANCTISGAAAQTAAASSLHQASNHTFYPWMAIAGECPEDPTKSKIRSDLTQYGGISTDMGKRYSESLAGSLLPDWLGTNGLRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHALCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAAAAAAAAAVQGGHLDQ
>Bombyx-mori_BGIBMGA006390-RA_NONE
MAHALCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAAAAAAAAAAAQGHPEH
>YOURSEQ3_Bombyx-mori_ultrabithorax
MNSYFEQGGFYGAHGVHQGGGGGDQYRGFPLGLTYAQPHALHQPRPQDSPYDASVAAACKLYAGEQQYPKADCSKPGGEQQNGYGGKEAWGSGLGALVRPAACTPEARYSESSSPGRALPWGNQCALPGSAASAAQPVHQQPTNHTFYPWMAIAGANGLRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHALCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAAAAAAAAAAAQGHPEH
>YOURSEQ5_Apis-mellifera_ultrabithorax
MNSYFEQTAGGFYGSHHHQTGAASQHHDPATAAAYRSFPLGLGMSPYASTQHHHHTSSSLGIHPGGGTNTRPPQDSPYDASVATACKLYSTTPEATGHTTSSYSTTAAKDCKQQDQASAHQNGYAAVMAAAAVKDVWQSATSGANSQSNSVVRPSACTPEGTRVGSYGGLVGGDPASSPGNNSSSRSLTSSWNTCSLNSSASQPVATQLHQQPSNHTFYPWMAIAGANGMRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHSLCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAAAAAAAAHQQQAAGGGPEGAN
>Apis-mellifera_GB51290-RA_UBX-Uncharacterized-protein
MNSYFEQTAGGFYGSHHHQTGAASQHHDPATAAAYRSFPLGLGMSPYASTQHHHHTSSSLGIHPGGGTNTRPPQDSPYDASVATACKLYSTTPEATGHTTSSYSTTAAKDCKQQDQASAHQNGYAAVMAAAAVKDVWQSATSGANSQSNSVVRPSACTPEGTRVGSYGGLVGGDPASSPGNNSSSRSLTSSWNTCSLNSSASQPVATQLHQQPTNHTFYPWMAIAGANGMRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHSLCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAAAAAAAAHQQQAAGGGPEGAN
>Bombus-impatiens_BIMP21073-RA_NONE
MNSYFEQTAGGFYGSHHHQTGAASQHHDPATAAAYRGFPLGLGMSPYASTQHHHHTSSSLGIHPGGGTNTRPPQDSPYDASVATACKLYSTTPEATGHTTSSYSTTATKDCKQQDQASAHQNGYAAVMAAAAVKDVWQSATSGSNSQSNSVVRPSACTPEGTRVGSYGGLVGGDPASSPGNNSSSRSLTSSWNTCSLNSSASQPVATQLHQQPTNHTFYPWMAIAGANGMRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHSLCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAAAAAPPIP
>YOURSEQ4_Bombus-impatiens_ultrabithorax
MNSYFEQTAGGFYGSHHHQTGAASQHHDPATAAAYRGFPLGLGMSPYASTQHHHHTSSSLGIHPGGGTNTRPPQDSPYDASVATACKLYSTTPEATGHTTSSYSTTATKDCKQQDQASAHQNGYAAVMAAAAVKDVWQSATSGSNSQSNSVVRPSACTPEGTRVGSYGGLVGGDPASSPGNNSSSRSLTSSWNTCSLNSSASQPVATQLHQQPTNHTFYPWMAIAGKEDIAKPHWTWLQGKRSARS
>Bombyx-mori_BGIBMGA006389-RA_NONE
MNSYFDQGGFYGAHGVHQGAAGGDQYRGFPLGLTYAQPHALHQPRPQDSPYDASVAAACKLYAGEQQYPKADCSKPGGEQQNGYGGKEAWGSGLGALVSPAACTPEARYSESSSPGRALPWGNQCALPGSAASAAQPVHQQPTNHTFYPWMAIAG
>YOURSEQ1_Heliconius-melpomene_ultrabithorax
AGEQQYAKADCSKAGGEQQNGYGGKEAWGSGLGALVRPAACTPEARYSESSSPGRALPWGNQCGLPGAAASAQPVQHQPTNHTFYPWMAI
>YOURSEQ2_Danaus-plexippus_ultrabithorax
MNSYFEQGGFYGAHGVHQGGGGGDQYRGFPLGLTYAQPHALHQPRPQDSPYDASVAAACKLYAGEQQYAKADCSKAGDQQNGYGGKEAWGSGLGALVRPAACTPEARYSESSSPSRALPWGNQCALPGAAASAQPVQHQPTNHTFYPWMAIAECELDTKEKIVSY
>Danaus-plexippus_DPOGS210960-TA_NONE
MNSYFEQGGFYGAHGVHQGGGGGDQYRGFPLGLTYAQPHALHQPRPQDSPYDASVAAACKLYAGEQQYAKADCSKAGDQQNGYGGKEAWGSGLGALVRPAACTPEARYSESSSPSRALPWGNQCALPGAAASAQPVQHQPTNHTFYPWMAIAECELDTKEKIVSY
>Heliconius-melpomene_HMEL025056-RA_Ubx-Ultrabithorax
MNSYFEQGGFYGAHGVHQGGGGGDQYRGFPLGLTYAQPHALHQPRPQDSPYDASVAAACKLYAGEQQYAKADCSKAGGEQQNGYGGKEAWGSGLGALVRPAACTPEARYSESSSPGRALPWGNQCGLPGAAASAQPVQHQPTNHTFYPWMAIA
>Heliconius-melpomene_HMEL025034-RA_Ubx-NONE
CFLFLFVSGANGLRRRGRQTYTRYQTLELEKEFHTNHYLTRRRRIEMAHALCLTERQIKIWFQNRRMKLKKEIQAIKELNEQEKQAQAQKAAAAAAAAAAAQGHPEH
>Bombyx-mori_BGIBMGA006388-RA_NONE
MIKVGPNGCPRRRGRQTYTRFQTLELEKEFHFNHYLTRRRRIEIAHALCLTERQIKIWFQNRRMKLKKELRAVKEINEQARREREEQDRMKQQQQEKQAKLEGQHHGHHVTHHHDPMKMPIDKGSNDLLKVNKVPT
>Drosophila-melanogaster_FBtr0083388_abd-A-abdominal-A-DBNLI
MSKFVFDSMLPKYPQFQPFISSHHLTTTPPNSSSAAVAAALAAAAASASASVSASSSSNNNSSNTIAGSNTSNTNNSSSSPSSSSNNNSNLNLSGGSLSPSHLSQHLGQSPHSPVSSSSPFQQHHPQVQQQHLNHQQQQHLHHQQQQHHHQYSSLSAALQLQQQQHHISKLAAAAVASHGHAHQQLLLTPPSAGNSQAGDSSCSPSPSASGSSSLHRSLNDNSPGSASASASASAASSVAAAAAAAAAAASSSFAIPTSKMYPYVSNHPSSHGGLSGMAGFTGLEDKSCSRYTDTVMNSYQSMSVPASASAQFAQFYQHATAAASAVSAASAGAIGVDSLGNACTQPASGVMPGAGGAGGAGIADLPRYPWMTLTDWMGSPFERVVCGDFNGPNGCPRRRGRQTYTRFQTLELEKEFHFNHYLTRRRRIEIAHALCLTERQIKIWFQNRRMKLKKELRAVKEINEQARRDREEQEKMKAQETMKSAQQNKQVQQQQQQQQQQQQQQQQQHQQQQQQPQDHHSIIAHNPGHLHHSVVGQNDLKLGLGMGVGVGVGGIGPGIGGGLGGNLGMMSALDKSNHDLLKAVSKVNS
>Acyrthosiphon-pisum_ACYPI009147-RA_NONE
MSSKFIIDSMLPKYHQQYHHQLLNPVITSGGGLDSSAVNYSLTPNNSSSSSAGGSPPSTSSLVSPAAGRMYPAYAVHHHQQQFGSGAGSMAFTSPGSAALAAAVQDAADKTSSSCRYGGGVGGVSVGGVSAADTMVNSYAALHHHHNQNGAGGTGAVSSMAAAAQFYHQAAAASAVAADPLNSACAGQPGAPGPQPMPDIPRYPWMSITDWMSPFDRVVCGPNGCPRRRGRQTYTRFQTLELEKEFHFNHYLTRRRRIEIAHALCLTERQIKIWFQNRRMKLKKELRAVKEINEQARRERDEQAKKQDHTKMDVGGGGGGMHQQQQQQQQQQQQQQQQQMSHHQMSHEQHKLQMGGLDKGSADMLKAKT
>Apis-mellifera_GB51287-RA_ABD-A-Homeobox-protein-abdominal-A
MSSKFIIDSMLPKYHQQFHHQQLFSSANPGTIQACTTSPATASLESSLSAAAVAAAAVNYAQQHNSPSPTGSSPQHSGSSASTSPAARTTSSMYPYVSAAAAHHHHQQQQAVAAAAFGATSSMVPGFGSTAASSAALAAAAAVDAATAGDKSCRYTASLAGNVAPASADPMVNYTLGHHHQNGATPSSLVSSASASSAVSAASASMAAAAQFYHQAAAASAVVDPLTSCQQPTTGQPSISDIPRYPWMSITDWMSPFDRVVCGPNGCPRRRGRQTYTRFQTLELEKEFHYNHYLTRRRRIEIAHALCLTERQIKIWFQNRRMKLKKELRAVKEINEQARREREEQEMMKKQQAEKQAKMQQEQQNAALQHQQQHHGVSGLDKTQSDLLKAVSKVPT
>Bombus-impatiens_BIMP20576-RA_NONE
MSSKFIIDSMLPKYHQQFHHQQLFSSANPGTIQACTTSPATASLESMNYAQQHNSPSPTGSSPQHSGSSASTSPAARTTSSMYPYVSAAAAHHHHQQQQAVAAAAFGATSSMVPGFGSTAASSAALAAAAXXXXXXXATAGDKSCRYTASLTGNVAPTSADPMVNYTLGHHHQNGATPGSLVSSASASSAVSAASASMAAAAQFYHQAAAASAVVDPLTSCQQPTTGQPGISDIPRYPWMSITDWMSPFDRVVCGPNGCPRRRGRQTYTRFQTLELEKEFHFNHYLTRRRRIEIAHALCLTERQIKIWFQNRRMKLKKELRAVKEINEQARREREEQDMMKKQQAEKQAKIQQEQQNAALQHQQQHHVSGLDKTQSDLLKAVSKVPT
>Danaus-plexippus_DPOGS210959-TA_NONE
MVNYALGQHNGGAAVSAASASMAAAAQFYHQAAASAASAASAASVDAMGAACSQPSAQPLPDIPRYPWMSITDFPFPDWMSPFDRVVCGEFNGPNGCPRRRGRQTYTRFQTLELEKEFHFNHYLTRRRRIEIAHALCLTERQIKIWFQNRRMKLKKELRAVKEINEQARREREEQDRMKQQQQEKQAKLESQHHGHHVTHHHDPMKMPIDKGSSDILKVNKVPT
>Heliconius-melpomene_HMEL025033-RA_abdA-NONE
MVNYALGQHNGGAAVSAASASMAAAAQFYHQAAASAASAASAASVDAMGAACSQPSAQPLPDIPRYPWMSITDWMSPFDRVVCGPNGCPRRRGRQTYTRFQTLELEKEFHFNHYLTRRRRIEIAHALCLTERQIKIWFQNRRMKLKKELRAVKEINEQARREREEQDRMKQQQQEKQAKLESQHHGHHVTHHHDPMKMPIDKGSSDILKVNKVPT
>Apis-mellifera_GB51292-RA_ANTP-Uncharacterized-protein
MSSYFANSYIPDLRNGGVEHPHQHQQHYGAAVQVPQQTQSVQQQSQQAGDPCDPSLLRQGVPGHHYGAAGSQQDMPYPRFPPYNRMDMRNATYYQHQQDHGSGMDGMGGYRSASPSPGMGHMGHTPTPNGHPSTPIVYASCKLQAAAVDHQGSVLDGPDSPPLVESQMHHQMHTQHPHMQPQQGQHQSQAQQQHLQAQQQHMMYQQQQQSQAASQQSQPGMHPQQQQQAQQHQGVVTSPLSQQQQAAPQGAASANLPSPLYPWMRSQFERKRGRQTYTRYQTLELEKEFHYNRYLTRRRRIEIAHALCLTERQIKIWFQNRRMKWKKENKSKGTPGSGDGDTEISPQTSPQG
>Bombus-impatiens_BIMP21075-RA_NONE
MSSYFANSYIPDLRNGGVEHPHQHQQHYGAAVQVPQQTQSVQQPSQQTGDPCDPSLLRQGVPGHHYGAAGSQQDMPYPRFPPYNRMDMRNATYYQHQQEHGSMDGLGGYRSTSPSPGMGHMGHTPTPNGHPSTPIVYASCKLQAAAVDHQGSVLDGPDSPPLVESQMHHQMHSQHPHMQPQQSQHQQQQQQHQHLQAQQQHMMYQQQQQTQAASQQSQPGMHPQQQQQPQQHQGVVTSPLSQQQQAAPQGAATANLPSPLYPWMRSQFERKRGRQTYTRYQTLELEKEFHFNRYLTRRRRIEIAHALCLTERQIKIWFQNRRMKWKKENKXGEPGSGDGDTEISPQTSPQG
>Drosophila-melanogaster_FBtr0081647_Antp-Antennapedia-DBNLI
MTMSTNNCESMTSYFTNSYMGADMHHGHYPGNGVTDLDAQQMHHYSQNANHQGNMPYPRFPPYDRMPYYNGQGMDQQQQHQVYSRPDSPSSQVGGVMPQAQTNGQLGVPQQQQQQQQQPSQNQQQQQAQQAPQQLQQQLPQVTQQVTHPQQQQQQPVVYASCKLQAAVGGLGMVPEGGSPPLVDQMSGHHMNAQMTLPHHMGHPQAQLGYTDVGVPDVTEVHQNHHNMGMYQQQSGVPPVGAPPQGMMHQGQGPPQMHQGHPGQHTPPSQNPNSQSSGMPSPLYPWMRSQFGKCQERKRGRQTYTRYQTLELEKEFHFNRYLTRRRRIEIAHALCLTERQIKIWFQNRRMKWKKENKTKGEPGSGGEGDEITPPNSPQ
>Acyrthosiphon-pisum_ACYPI008076-RA_NONE
MSSYFANSYMPSDMRNGGGGGTGGGLVGGGSGGGGVLGGVGGIVGGGGGGGGGVVMDHHHHGQQHYVDNAPCDPTARQGIPPHHYVGPTVGGQPPQGMPYPRFPPYDRMEIRQAAAAAGYYNNHHQSSMDGYSRPESPIGGGLVGGGGGGGGGMGQMTMNGHTPVVYATCKLQASVANGLVEPGSPTDMVDMTNHHQQQQQQQQQMTAAGHHHHQQQQHHHSAPPPQSHHQQQQQAYFSNLSPHHHQQQQQQQQSVVPPAPGHHNQVVVNPHQQTPPLQTPTAQNQQAVNNNSLPSPLYPWMRSQFERKRGRQTYTRYQTLELEKEFHFNRYLTRRRRIEIAHALCLTERQIKIWFQNRRMKWKKENKTKGGDGQGGDGSDITPQTSPQ
Arbre des espèces en format Newick
((((Heliconius-melpomene_ultrabithorax:0.04746, Danaus-plexippus_ultrabithorax:-0.00252):0.10323, Bombyx-mori_ultrabithorax:-0.04691):0.18054, (Bombus-impatiens_ultrabithorax:0.08315, Apis-mellifera_ultrabithorax:-0.00592):0.18318):0.01668, Acyrthosiphon-pisum_ultrabithorax:0.17923, Drosophila-melanogaster_ultrabithorax:0.17042);
Arbre de genes en format Newick
((((Acyrthosiphon-pisum_ACYPI008076-RA_NONE:0.069959,Drosophila-melanogaster_FBtr0081647_Antp-Antennapedia-DBNLI:0.067967)21:0.011595,(Bombus-impatiens_BIMP21075-RA_NONE:0.000741,Apis-mellifera_GB51292-RA_ANTP-Uncharacterized-protein:0.029014)95:0.067917)12:0.013428,((((((((Heliconius-melpomene_HMEL025033-RA_abdA-NONE:0.000000,Danaus-plexippus_DPOGS210959-TA_NONE:-0.000000)100:0.068080,((Bombus-impatiens_BIMP20576-RA_NONE:-0.001001,Apis-mellifera_GB51287-RA_ABD-A-Homeobox-protein-abdominal-A:0.010750)99:0.062209,Acyrthosiphon-pisum_ACYPI009147-RA_NONE:0.052330)49:0.014235)82:0.009336,Drosophila-melanogaster_FBtr0083388_abd-A-abdominal-A-DBNLI:0.113075)61:0.021270,Bombyx-mori_BGIBMGA006388-RA_NONE:-0.060358)88:0.072036,(Heliconius-melpomene_HMEL025034-RA_Ubx-NONE:-0.033603,(((((((((((Heliconius-melpomene_HMEL025056-RA_Ubx-Ultrabithorax:-0.376936,(Danaus-plexippus_DPOGS210960-TA_NONE:-0.000000,YOURSEQ2_Danaus-plexippus_ultrabithorax:0.000000)39:0.413676)7:0.196399,YOURSEQ1_Heliconius-melpomene_ultrabithorax:-0.195614)49:0.155196,Bombyx-mori_BGIBMGA006389-RA_NONE:-0.079621)49:0.166028,YOURSEQ4_Bombus-impatiens_ultrabithorax:0.346982)89:0.447706,Bombus-impatiens_BIMP21073-RA_NONE:-0.266426)2:0.154751,Apis-mellifera_GB51290-RA_UBX-Uncharacterized-protein:-0.139552)5:0.083488,YOURSEQ5_Apis-mellifera_ultrabithorax:-0.066029)5:0.104873,YOURSEQ3_Bombyx-mori_ultrabithorax:-0.007868)22:0.024514,Bombyx-mori_BGIBMGA006390-RA_NONE:-0.056931)31:0.011877,(Drosophila-melanogaster_FBtr0083347_Ubx-Ultrabithorax-DBNLI:0.000000,YOURSEQ7_Drosophila-melanogaster_ultrabithorax:0.000000)96:0.056809)7:0.022825,(Acyrthosiphon-pisum_ACYPI001856-RA_NONE:0.000000,YOURSEQ6_Acyrthosiphon-pisum_ultrabithorax:-0.000000)100:0.075773)65:0.028070)62:0.120899)67:0.053125,Bombyx-mori_BGIBMGA006393-RA_NONE:0.316543)21:0.030914,(Danaus-plexippus_DPOGS210965-TA_NONE:-0.050592,(Apis-mellifera_GB51295-RA_SCR-Homeobox-protein-H55:0.095928,Acyrthosiphon-pisum_ACYPI37793-RA_NONE:0.099688)82:0.017135)80:0.103898)42:0.069500,Bombyx-mori_BGIBMGA006392-RA_NONE:0.029599)62:0.027276)5:0.017833,Danaus-plexippus_DPOGS210962-TA_NONE:-0.051894,Heliconius-melpomene_HMEL025000-RA_Antp-NONE:0.051894);
Résultats obtenus suite à l’analyse sur T-Rex